人生で初めて[海外に||一人で||猫と]暮らしている(現在進行系)
いい機会なので、滞在中の心境を研究留学 Advent Calendar 2017に似せたフォーマットで纏めておきます。
どうやって行ったか
8月11日、研究室の指導教員から「ベルギーにいる僕の友人が、短期滞在で研究員を探しているんですけど、興味ありますか?」と言われた。
自分が所属しているコースでは、課程を卒業するために日本国外で3ヶ月以上の研究留学、あるいはインターンシップに参加することが求められている。一方でこのコースに入っていると、特定の条件下で生活費が支給される。自分は元々留学には興味がなかったが、そちらの生活費が目当てで加入した。
ただ、その後別途生活費は確保することが出来たので、必ずしも海外に留学する必要は無くなった。何故ならこのコースは脱退罰則が定義されていない。所属することの恩恵は月々の収入や外部試験と出張の補助であるが、学振DC1を取っている場合これらの補助は殆ど自身で賄うことが出来る。なので留学が面倒ならば、課程自体を辞めてしまえばよい。
しかし海外で生活するということは自分にとっては全くの新体験。将来的に仕事でこういう機会は訪れるかもしれないが、Webを見ていると若いうちに海外での生活を経験しておくことは多くの収穫があると述べる文章は多い。
一方で、留学をすることで学位取得が遅れる可能性はある。しかしそのリスクを踏まえた上でも、長期的に見れば意味があるのかもしれないと考えて行くことを決めた。留学するならば、コースから補助が出るわけだし、経済的に損はない。そういうわけで、渡航費用*1と滞在費*2については負担が無い。ただしその他の生活費については自腹だった。
どこに行ったか (組織など)
ベルギーのKU Leuvenあるいは、VIBへ行った。滞在先のボスの正式な所属はVIB-KU Leuven Centerであり、自身のIDカードもKU Leuvenから発行されている。研究室もベルギー北部のLeuven市に存在する。しかし実質的な研究室ポジションはVIB寄りである。
VIB
VIB(Vlaams Instituut voor Biotechnologie)については邦語では殆ど情報がない。なお日本語の資料だと、東工大の調査レポートが分かりやすい。
VIBについて語るためには、まずベルギーについて語る必要がある。この国はチョコレート、小便小僧、国連本部というイメージで語られることが多い*3。実際にはヨーロッパ中でのポジショニングとしては複雑な国である。
九州程度の国土なのだが、その中でオランダ語圏、フランス語圏、そして少々のドイツ語圏が存在する。オランダ、イギリス、フランス、ドイツに囲まれるベルギーは古くからヨーロッパの交易の要所として栄えた。元々はオランダ語が優勢な国であったのだが、ナポレオンの統治の際にフランス語が公用語として使われた結果、国内のフランス語圏勢力が拡大する*4。一方で庶民の生活を描くことに重きをおいたロマン主義運動や、ポストナポレオンのパワーバランスの為に非フランスとしてのオランダが重視された結果オランダ語圏も力を持った。この結果としてフランス語圏とオランダ語圏が入り混じって存在するベルギーが生まれたのだとか。詳細は以下の新書を参考にして欲しい。

- 作者: 松尾秀哉
- 出版社/メーカー: 中央公論新社
- 発売日: 2014/08/22
- メディア: 新書
- この商品を含むブログ (10件) を見る
以上の経緯のために、国内では2つの勢力が独自に存在する。VIBはそのうち、オランダ語圏のフランデレン政府によって作られた生物学系の研究所だ。 面白いのは、VIBそのものは研究所を保たないことである。フランデレン地方の大学と提携・資本協力することでそれぞれの大学に講座を構えている。この機関のもう一つの特徴は、産業創出を重視している点である。財源の少なくない割合を技術移転にあてており、実際に幾つかのベンチャー企業も誕生しているんだとか。VIBの年額の予算は2017年から5年かけて増額され5900万ユーロ程度(=80億円)まで引き上げられるらしい。これは大学の額と直接比較すると多くはない*5。注意するべきなのは、VIBはあくまで生命科学の研究所だと言う点だ。ここで比較するべきは全体額ではなくLife scienceの分野との比較であり、それだとMITでも1.29億ドル(=140億円)、大学平均だと68億円だ。しかもVIBの研究室は所属大学からの恩恵も受けられる。予算額としては中々良い研究機関である。
なぜそこへ行ったか
1番大きな理由はボスがその研究室のボスの元同僚だったからだ。知己があると行きやすい。ラッキー。
しかしもう1つ、滞在先のパフォーマンスに興味を持った。滞在先のボスは2017年度は30報の著者論文を書いた。この内インパクトファクターが10を上回る雑誌から出版されたものが12報あり、その内Natureへ3報、それとは別にNature系列へ3報出ている。個人的な感想を言えばモンスターだった。すごい。そのような環境で研究をすることで、パフォーマンスの秘密を知りたかった。
いつ行ったか
短期の滞在で、2017/12/01 ~ 2018/02/28まで滞在する予定で。ちょうど今日が折り返し地点というわけです。より具体的なスパンとしては、
- 6月: 滞在先のボスが来日し、カンファレンスで発表する。
- 8月: 声がかかる
- 9~10月: 日程調整
- 10月下旬: 日程決定
- 12月: 出発
といった流れだった。
このような日程になったのは3つの思惑が複雑に絡み合った結果と言えよう。
1つ目は、自身の考えである。自分は滞在によって得られる経験値と、自身の研究が遅れるトレードオフを踏まえると、単位として最低限必要な3ヶ月がベストであった。 特に後者の要因が非常に大きい。自分の所属している大学院の課程では、学位を取るために指導教員が著者に入っている(あるいは単著の)筆頭著者の査読付き論文と指導教員の合意が必要である*6。自分の場合だと、「指導教員が著者に入った」という点が重要である。この結果として、留学先に長期滞在して論文を発表したとしても、卒業要件の論文としてはカウントされない。そのため滞在すれば滞在するだけ、自身の研究は進まず卒業が難しくなる。
更に、自分の所属している大学院では、学位審査の委員会に指導教員が入っている。この制度は中々独特らしい。こちらの留学を妨げる要因となった。仮に読者がそこそこの貢献をした(指導教員も著者に入っている)論文を発表したとする。この場合でも指導教員の胸先一寸で学位取得が左右され、学位取得は安泰ではない。委員会に指導教員が入っているのだから、彼の人が持つ決定権が大きいのだ*7*8。自分もリスクを考え、留学に来る前に日本の指導教員とこの点は話した*9。しかしながら彼の人曰く、「留学先で査読付き論文が出ても、それで学位をあげるつもりは無い」そうだ。 以上の理由から、自分が長期的に滞在することは、学位取得の点ではまるで得が無い*10。よっていいとこ取り*11で3ヶ月を希望した。
2つ目は、滞在先のボスの思惑である。自分の研究領域はバイオインフォマティクスで基本的には生命科学*12なので、3ヶ月では論文出版まで漕ぎ着けることが難しい。そのためより長期の滞在が期待され、国内のボスも混じえた日程調整に時間がかかって12月~2月の滞在となった。この日程だとビザを用意する必要もなく、色々と楽である。
3つ目は、予算機関の思惑である。自分の所属しているコースの予算は今年度末が期限だった。そのため、予算が満額支給されるためには、年度内に帰国する必要があった。
何をやっているか
当たり前だが守秘義務があるので、大した事は言えない。が、相変わらずバイオインフォマティクスをやっている。しかし元々自分は(これまでのブログ記事を見れば分かるように)統計学寄りで、塩基配列そのものはあまり触ってこなかった*13。今は塩基配列を触っており、BLASTやらSAMTOOLSやらSEQ-KITなどのツールを勉強する良い機会になった。
自分は外の釜の飯を食うことも重要だと思う。特定のテーマについて追求するためには、間違いなく継続的に続けることは重要だ。その点では留学は重荷でしか無い。一方で、特定のテーマについて掘り進める方法は色々ある。自分の研究室から離れて、異なるアプローチを知るためには武者修行が良い手段である。
何を知りつつ有るか
まだ現在進行系だが、色々と勉強になることが多い。依然わからないことも多い。
労働時間
よく言われることだが、こちらの人々は遅くまで働かない。大体17時ぐらいには帰り始め、18時ともなると殆どおらず、19時には研究室が真っ暗だ*14。予想はしていたが、長く労働する傾向の有る自分には衝撃的であった。とはいいつつも総労働時間が極端に短いわけでもない。朝来る時間はそこそこ早く、9時には結構な割合が来ているし、8時に来る人もいる。残業が無いと踏まえるのが適当だ。
滞在先のボスにこのことを訊いたが、結局帰ってからも仕事をしている人が多いらしいので、パフォーマンスの確実なミラクルは存在しないらしい。彼が述べていた興味深い仕事は、メンバーの忙しさを管理しているという事だ。仕事の中にも重い軽いはあるので、それを見極めて常に過負荷にならないように調整しているらしい。マネージャーともなれば当然なのかもしれないけれども、ラボのPIが単純に仕事を降るだけではなく、調整弁までこなしているのは安心することができよう。
ただまぁ、クリスマスはしっかりと休んでいた。自分の研究のメンターは12/20ぐらいから休暇を取り始め、1/5ぐらいまで現れなかった。研究所自体も12/23から1/1まで閉鎖された。
思うに彼らはメリハリの付け方が上手い。自分だと家に仕事を持ち帰りたくないので、研究室に滞在する時間が長くなりがちだ。スイッチングを調整できていることは、パフォーマンスの源泉の1つだろう。しかし、そのマインドの習得方法は未だにわからない。
ポジション
これもよく言われることだが、サポーティングスタッフが多い。所謂Lab technicianと呼ばれる。
- Prof: 1
- Post Dr. researcher: 5~6
- Technician: 5~6
- Doctor course student: 5~6
- Master course student: 3~4?
ぐらいだろうか。これには秘書などの研究に関わらないメンバーは含めていない。更に学部生も居ない。そのため教育のためにかかるコストは日本と比べると低いように思える。
一方で、自分のイメージとは異なり、ポスドク研究者がチームをリードすることも多い。自分はこのような仕事は助教以上のタスクかと思っていたが、よりElderなポスドクには任されて責任著者へ繋げるらしい。とはいえ、これは研究室に依るのだろう。
VIBそれ自体でも、Techに対するサポートは篤い。VIBは様々なトレーニングコースを用意しており、自由に参加することも出来る。その中にはTechのみを対象としたものが数多く存在するし、キャリアについて考えるものが専用に用意されている。日本では研究をサポートするURA*15やTechが全然居らず、教員へ仕事が回ることでの負担が取り沙汰されるが、このような基盤まで固めるのは一朝一夕では無理だろう。
コミュニケーション
研究室のベルギー人率は50%程度で、内部の公用語は専ら英語だ。なので特に困ることはない。しかし行ってみるまで知らなかったのだが、研究室メンバーの女性の割合が非常に高い。男女比は3:7ぐらいだ。
そのためなのか、お昼ごはんを一緒に食べることが半ば1つの義務みたいになっている。この点は少しびっくりした。日本は集団文化であり、欧米は個人主義とは聞いたものだが、全く逆の状態であった。むしろ1人で食事をしていると心配されることが多い。少なくとも調べた所、フランスでは(カフェなどのフランクな場は除く)外食を1人でする文化はないと出てくるし、単に欧米文化の特徴なのかもしれない。だとすれば日本人が考えている以上に、学会なんかのレセプションに出席して親交を深めることは重要な事項なのだろう*16。同室の学生の言うところによると「食事は社会活動*17なんだから一緒に食べないとダメでしょ」ということだ。
一人暮らしと猫
日本ではずっと実家ぐらしだったので、一人暮らしに悪戦苦闘している。
- ヨーロッパのキャベツは固い。調理に困った。
- 出汁をなくして、初めて重要さに気がついた。
- 表示がオランダ語/フランス語なので、野菜はともかく、肉は全く分からない。
だがしかし、下宿先に猫が居り、今まで叶わなかった猫との生活が達成できた。僥倖である。

物凄く嫌がられている気がしなくもない。
結び
こちらの滞在も半分を切った。なんとかハイパフォーマンスの方法論の欠片でも掴みたい。
履歴
2018-01-17: どうやって行ったか、の題と文が合ってなかったので追記 2018-01-30: 読み返して恥ずかしくなった部分を削除
*1:自宅から空港+往復航空券+空港から下宿先
*2:家賃+研究室までの定期券
*3:自分もそんなイメージだった
*4:特に当時のエリートはフランス語を用いたため、高級階層はフランス語がメインだったんだとか。
*6:個人的な物言いだが、博士課程への警告は山ほど目にし耳に入ってくるものの、卒業要件について確認するべきとの声を聞いたことはない。この基準は大学毎、時により研究科毎にかなり異なる。機関によっては査読付き論文が必要がないところもあるし、逆に3報の筆頭著者論文(化学系など)が必要な場合もある。もし読者が博士課程に進学する際には、ゆめゆめ確認して欲しい。
*7:このような状況にあり、学位が取得できていない学生は研究科に複数実在する。
*8:逆に指導教員の決定次第で学位取得がかなり容易にもなりうる。しかし分散が大きいだけなので品質保証としてはやはり良くない
*9:なんでも指導教員も自身がこちらで取り組んでいる研究には関わっているらしく著者に入るらしい
*10:勿論研究者として生きていくことを考えるなら、別である。良い論文を出せる確率がある。
*11:中途半端ともいう
*12:bioinformaticsだと研究成果について生物学的な知見が要る。そのため大体1~2年かかるイメージだ。一方でcomputational biologyだと計算機科学の観点が強く、上手く行けばこの短期でも論文になろう。
*13:なので自分の専門はbiostatistics、あるいはtablomicsかdatabasomicsなどと述べていた
*14:同室の学生は「金曜日には16:30でも帰るには遅すぎる」という名言を残した
*15:University Research Administrator。日本でのURAの働きは、遺伝研の要覧が分かりやすい。獲得したグラントや出版論文が丁寧に纏めてあり機関の重要性がよく伝わる。
*16:現状の日本の制度では、懇親会費は個人の自腹となっている
*17:social activity
Stanで生存時間解析(Weibull 回帰)
生存時間解析とは?
生存時間解析は、イベントの時間を解析するための手法です。例えば、
- ソーシャルゲームやwebサービスなどに登録した人の利用継続時間(マーケティング)
- 投薬群と対照群(プラセボ)で、どれだけ長生きするか(医用統計)
- 新規材料が既存の材料とくらべて、どれぐらい耐久性があるか(信用工学)
など、様々な応用例が考えられます。生存時間解析はノンパラメトリックな手法で行なうことが多いです。具体的には、
- カプランマイヤー推定量で生存時間を推定
- 生存時間の違いをログランク検定で判断
- 共変量の違いをCoxの比例ハザードモデルやAelanの加法モデルで解析
といった辺りが挙げられるでしょうか。その原因としては、生存時間のデータへ打ち切りという特徴があることが挙げられます。結果として分布にあてはめるだけの解析では、打ち切りを説明することが難しく上手くフィッティング出来ません。並べて、最尤法を使った分布への当てはめでは、値の信頼性が出ません。これに対してノンパラメトリックなカプランマイヤー法を使えば、推定値の範囲(信頼性)も出せますので論文も書きやすいです。
しかし仮に分布を仮定しても、ベイズ的にモデリングすれば、これらの問題を解決することができます。今回はStanを使って生存時間解析を行なうことで、打ち切りを含めてモデリングをしてみました。何分不勉強なもので、ご指導ご鞭撻をいただければ幸いです。
テストデータ
とりあえず解析に使うデータを用意します。今回はpythonのlifeline packageに含まれる、rossiのデータを使います。データのソース論文はこちらです。このデータは逮捕された人が再逮捕されるまでの期間(週)と、その他の共変量が記されています。何はともあれ、データを見てみましょう。上から10件だけ表示します。
from lifelines.datasets import load_rossi df = load_rossi() df
| index | week | arrest | fin | age | race | wexp | mar | paro | prio |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 20 | 1 | 0 | 27 | 1 | 0 | 0 | 1 | 3 |
| 1 | 17 | 1 | 0 | 18 | 1 | 0 | 0 | 1 | 8 |
| 2 | 25 | 1 | 0 | 19 | 0 | 1 | 0 | 1 | 13 |
| 3 | 52 | 0 | 1 | 23 | 1 | 1 | 1 | 1 | 1 |
| 4 | 52 | 0 | 0 | 19 | 0 | 1 | 0 | 1 | 3 |
| 5 | 52 | 0 | 0 | 24 | 1 | 1 | 0 | 0 | 2 |
| 6 | 23 | 1 | 0 | 25 | 1 | 1 | 1 | 1 | 0 |
| 7 | 52 | 0 | 1 | 21 | 1 | 1 | 0 | 1 | 4 |
| 8 | 52 | 0 | 0 | 22 | 1 | 0 | 0 | 0 | 6 |
| 9 | 52 | 0 | 0 | 20 | 1 | 1 | 0 | 0 | 0 |
| 10 | 52 | 0 | 1 | 26 | 1 | 0 | 0 | 1 | 3 |
詳細は論文の3.2節に書いてありますが、今回注目するのはweekとarrestです。arrestが1である人は再逮捕されています。0である人は観測期間中では再逮捕はされず、weekの値が52になっています。所謂打ち切りの発生です。
モデル
アヒル本P115 7.8 打ち切り、に従い、現象をモデル化してみます。なお今回のデータはweek単位への丸めも発生しているのですが、本題ではないので飛ばします。
打ち切りの原理を踏まえて、今回の現象のモデルを次のように考えました。
「再逮捕の区間を表すパラメータと分布に従って、再逮捕までの時間が得られる。ただし
である場合は、
とする 」
分布はワイブル分布を利用します。ワイブル分布は次のような特徴をもち、時間のモデル化へ向いています。
における累積密度関数の和が1になる(イベントの発生確率を無限時間まで考えられる)
- 現象の傾向を示す形状パラメータ
と、現象の時間スケールを示す尺度パラメータ
で表現される
なら、時間とともにハザード関数値(時間
までにイベントが発生しなかった場合、その瞬間にイベントが起こる確率)が低くなる
なら、時間に依らずハザード関数値が一定
なら、時間にとともにハザード関数値が高くなる
ワイブル分布を使って、値に対する尤度を考えます。観測値が52で無い場合は
として得られます。肝心なのは52の場合です。この時尤度は
となります。
以上を踏まえて、まず共変量に依らず、全ての人が共通の現象に従っているとして、Stanを書いてみました。
data {
int N ;
int week[N] ;
int arrest[N] ;
}
parameters {
real shape ;
real scale ;
}
model {
for(n in 1:N){
if(arrest[n] == 0){
target += weibull_lccdf(week[n]| shape, scale) ;
}else{
target += weibull_lpdf(week[n]| shape, scale) ;
}
}
}
generated quantities {
real log_lik[N] ;
for(n in 1:N){
if(arrest[n] == 0){
log_lik[n] = weibull_lccdf(week[n]| shape, scale) ;
}else{
log_lik[n] = weibull_lpdf(week[n]| shape, scale) ;
}
}
}
解析と可視化
データに対して、構築したStanモデルを当てはめてみます。
import numpy as np import os import pystan import pandas as pd import pickle from lifelines.datasets import load_rossi def stan_compile(stan_file_path, compiled_file_path, recompile=False): if os.path.exists(compiled_file_path) is False or recompile is True: model = pystan.StanModel(file=stan_file_path) with open(compiled_file_path, "wb") as f: pickle.dump(model, f) else: with open(compiled_file_path, "rb") as f: model = pickle.load(f) return model def get_summary_df(fit): summary = fit.summary(probs=prob) summary_df = pd.DataFrame(summary["summary"], index=summary["summary_rownames"], columns=summary["summary_colnames"]) return summary_df def get_waic(fit): log_lik = fit.extract("log_lik")["log_lik"] waic = (-2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0))) return waic # Stanに入れるデータの準備 df = load_rossi() N = len(df) week = df["week"] arrest = df["arrest"] stan_data = {"N": N, "week": week, "arrest": arrest} prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975] # Stanをコンパイル stan_file = os.path.join("weibull.stan") stan_file_c = os.path.join("weibull.pkl") model = stan_compile(stan_file, stan_file_c) # StanへKick fit = model.sampling(data=stan_data, iter=3000, chains=5, warmup=1000, seed=1234) # 結果の集計 summary_df = get_summary_df(fit) waic = get_waic(fit) print(waic) summary_df
output
WAIC: 1397.22843377
| parameter | mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| shape | 1.35177 | 0.00225134 | 0.123002 | 1.12027 | 1.15716 | 1.26817 | 1.347 | 1.43093 | 1.56233 | 1.61164 | 2985 | 1.00366 |
| scale | 127.559 | 0.267877 | 14.5396 | 103.759 | 106.833 | 117.305 | 125.893 | 136 | 153.639 | 161.079 | 2946 | 1.00274 |
| log_lik[0] | -5.26822 | 0.000938137 | 0.0870498 | -5.44863 | -5.41606 | -5.3248 | -5.26577 | -5.20816 | -5.13211 | -5.10419 | 8610 | 0.999675 |
| log_lik[1] | -5.30895 | 0.000968553 | 0.0933687 | -5.5044 | -5.46746 | -5.36939 | -5.30494 | -5.2457 | -5.16148 | -5.13464 | 9293 | 0.999847 |
| log_lik[2] | -5.21905 | 0.00110178 | 0.0852228 | -5.39228 | -5.36399 | -5.27516 | -5.21646 | -5.16062 | -5.08346 | -5.05936 | 5983 | 1.00011 |
| … | … | … | … | … | … | … | … | … | … | … | … | … |
まず集計表です。全パラメータでが確認できましたので、収束は問題ないようです。続いて、得られたパラメータ値を用いて可視化を行います。
from scipy.stats import weibull_min import matplotlib.pyplot as plt # 可視化その1 fig, ax1 = plt.subplots() x = np.arange(0, 500, 1) y = weibull_min.pdf(x, c=summary_df.loc["shape", "mean"], scale=summary_df.loc["scale", "mean"]) y_min = weibull_min.pdf(x, c=summary_df.loc["shape", "mean"], scale=summary_df.loc["scale", "5%"]) y_max = weibull_min.pdf(x, c=summary_df.loc["shape", "mean"], scale=summary_df.loc["scale", "95%"]) # 確率密度関数 ax1.plot(x, y, "-") ax1.fill_between(x, y_min, y_max, facecolor="pink") ax1.set_ylabel("probability") ax1.tick_params("y") ax1.set_xlabel("Weeks") # 件数 ax2 = ax1.twinx() ax2.hist(df["week"]) ax2.set_ylabel("count") ax2.tick_params("y") plt.show() # 可視化その2 fig, ax1 = plt.subplots() x = np.arange(0, 50, 1) y = weibull_min.sf(x, c=summary_df.loc["shape", "mean"], scale=summary_df.loc["scale", "mean"]) y_min = weibull_min.sf(x, c=summary_df.loc["shape", "mean"], scale=summary_df.loc["scale", "5%"]) y_max = weibull_min.sf(x, c=summary_df.loc["shape", "mean"], scale=summary_df.loc["scale", "95%"]) # 生存時間関数 ax1.plot(x, y, "-") ax1.set_ylabel("Propotion Not Arrested") ax1.fill_between(x, y_min, y_max, facecolor="pink") ax1.set_xlabel("Weeks") ax1.tick_params("y")


一枚目のグラフが、確率密度関数とイベント発生のカウントです。MCMCのEAPが青線、scale parameterの90%信用区間を用いて描かれた範囲がピンク色になっています。
二枚目のグラフが生存時間関数(survival function)です。見方は1枚目と同様です。元論文のP7には、Coxの比例ハザードモデルで描かれた場合の結果が載っています。今回のWeibull分布を用いて描いた結果と相違ありません。
打ち切り補正をしない場合は?
当然ここで気になってくるのは、補正の効果です。先程の1枚目のグラフは52辺りに最頻値があります。打ち切りをしている効果はどの程度でしょうか?これを検証するために、打ち切りを考慮しないモデルと比較しました。

打ち切りを考慮しない場合は、めちゃめちゃ過剰適合しているのがわかります。打ち切りのメカニズムをモデルに取り入れることで、52以上でもイベントが生起することを踏まえてモデル化できてるわけですね。ちなみに打ち切りが考慮されない場合、WAICも3411.15613467となり、モデルとしても性能が良くないことが分かります。
比例ハザードモデル
次に共変量の効果を調べます。一番単純な比例ハザードモデルで取り組んでみましょう。比例ハザードモデルは、全時間を通してハザード間数値を一定として、共変量に従ってハザード関数の値が大きくなることを仮定するモデルです。Weibull分布のハザード関数は
と示されます。先行研究[1][2]では、尺度パラメータへ共変量の効果を加えています。この理由の僕個人による定性的な理解としては、形状パラメータの頑強性に帰するものと考えています。形状パラメータの値はそもそもの現象に依存します。共変量ぐらいでは、中々現象の傾向までは大きく変わりません。
更に、尺度に対する効果も尺度パラメータへダイレクトに加えるのではなく、
へ加えるほうが多いようです。即ち基底状態に対するパラメータを
、共変量の係数を
、共変量のダミー変数を
として、
ということになります。この式をを中心に変形すれば
です。Stanで実装するときは、この式で書けばよいかと思います。
今回の解析では、逮捕後の経済的支援(financial aid)の効果を調べてみたいと思います。モデルは次の通りです。
data {
int N ;
int week[N] ;
int arrest[N] ;
int fin[N] ;
}
parameters {
real shape ;
real beta[2] ;
}
model {
for(n in 1:N){
if(arrest[n] == 0){
target += weibull_lccdf(week[n]| shape, exp(- (beta[1] + fin[n] * beta[2]) / shape)) ;
}else{
target += weibull_lpdf(week[n]| shape, exp(- (beta[1] + fin[n] * beta[2]) / shape)) ;
}
}
}
generated quantities {
real log_lik[N] ;
for(n in 1:N){
if(arrest[n] == 0){
log_lik[n] = weibull_lccdf(week[n]| shape, exp(- (beta[1] + fin[n] * beta[2]) / shape)) ;
}else{
log_lik[n] = weibull_lpdf(week[n]| shape, exp(- (beta[1] + fin[n] * beta[2]) / shape)) ;
}
}
}
スクリプトは殆ど同様なので飛ばします。結果として次のグラフが得られました。


2枚目のグラフが分かりやすいですね。経済的支援を受けた場合は、再犯率が低くなっています。WAICも1395.3474841となって低くなり、予測力が上がっています。更に言うと、経済的支援の効果を表すパラメータに対する、
の95%信用区間(片側)は
でした。この区間に1(即ち効果が無い)が入っていませんので、経済的支援に効果があったことが期待されます。
まとめ
Python + Stan + Weibull分布を使って、打ち切りを含めたモデル化を行い、共変量の効果の信用区間まで計算しました。…あれ、これはCoxの比例ハザードモデルで十分なのでは(死)。
ベイズ的にモデル化することの利点は自由性です。パッと思いつくものとして、次のような応用が挙げられます。
- 加法モデルやオッズ比モデルの利用
- 値の丸めを加味したモデル
- 時間tに対して、ハザード関数が変化するモデル(比例ハザードモデルでは一定とする)
- リンク関数の中身へ正規化項を追加して、スパースにする(Lasso + weibull 回帰?)
- 単純な2群比較のコホートデザインではない場合(利用できる共変量が他にある)
ふぅー、これでやった意味が見いだせました。ぜひ皆様も、Weibull分布に親しんで下さい。
参考
書籍

- 作者:豊田 秀樹
- 出版社/メーカー: 朝倉書店
- 発売日: 2017/01/25
- メディア: 単行本(ソフトカバー)
2節「ワイブル分布」がドンピシャでした。
打ち切りデータのモデリングについて勉強させて貰いました。
Webリソース
- Modeling Language User's Guide and Reference Manual, Version 2.16.0
- Cox Proportional-Hazards Regression for Survival Data in R
- COMPARISON BETWEEN WEIBULL AND COX PROPORTIONAL HAZARDS MODELS
- Estimating trends in data from the Weibull and a generalized extreme value distribution
- Lifelines
- 比例ハザードモデルはとってもtricky!
- Bayesian Lassoで特徴選択
StanとPythonでベイズ統計モデリング その3 Chapter6
アヒル本(StanとRでベイズ統計モデリング)のChapter6にPythonで取り組んでいきます。 この章は丁寧に分布を解説していくものなので、内容の復習は飛ばします。おざなりにされそうな章ですが、自分でパラメータをいじって分布からサンプリングしてみると新しい発見もありますので挑戦してみるのをおすすめします。
今回はRどころかStanも出てきません(笑)
練習問題
練習問題(1)
ベルヌーイ分布 or カテゴリカル分布に従う乱数を生成せよ…ということですが、numpyには両方とも存在しません。 二項分布からのサンプリングを一回だけ行うものがベルヌーイ分布であり、多項分布からのサンプリングを一回だけ行うものがカテゴリカル分布であるため、それで代用しました。
カテゴリカル分布についてはRの結果と対応させるために少し手間を加えます。
import numpy as np random_bernoulli = np.random.binomial(1, 0.5, 100) random_categorical = np.random.multinomial(1, [0.2, 0.3, 0.5], 100) np.where(random_categorical == 1)[1]
練習問題(2)
テキストで推奨されている、ベータ分布、ディリクレ分布、ガンマ分布、2変量正規分布、コーシー分布を生成していきます。ついでに可視化します。
ベータ分布
alphaが大きいほど右側に偏り、全体的に値が大きいほど真ん中(0.5辺り)に集合していくことが分かりました。
import numpy as np import matplotlib.pyplot as plt import seaborn as sns alpha = 0.5 beta = 0.5 r_beta = np.random.beta(alpha, beta, size=1000) g = sns.distplot(r_beta, kde=False, label="alpha={0}, beta={1}".format(alpha, beta)) alpha = 1 beta = 0.5 r_beta = np.random.beta(alpha, beta, size=1000) g = sns.distplot(r_beta, kde=False, label="alpha={0}, beta={1}".format(alpha, beta)) alpha = 1 beta = 1 r_beta = np.random.beta(alpha, beta, size=1000) g = sns.distplot(r_beta, kde=False, label="alpha={0}, beta={1}".format(alpha, beta)) alpha = 5 beta = 5 r_beta = np.random.beta(alpha, beta, size=1000) g = sns.distplot(r_beta, kde=False, label="alpha={0}, beta={1}".format(alpha, beta)) g.legend() plt.show(g)

ディリクレ分布
トピックモデルで使われる分布です。ベータ分布の多次元拡張なので、alphaに対する挙動はベータ分布と同じです。
今回は3次元で行いました(可視化の限界のためです…)。 可視化の方法は先行事例を参考にしています。
import matplotlib.tri as tri import numpy as np def plot_points(X, barycentric=True, border=True, **kwargs): ''' Copy from https://gist.github.com/tboggs/8778945 ''' corners = np.array([[0, 0], [1, 0], [0.5, 0.75**0.5]]) triangle = tri.Triangulation(corners[:, 0], corners[:, 1]) if barycentric is True: X = X.dot(corners) plt.plot(X[:, 0], X[:, 1], 'k.', **kwargs) plt.axis('equal') plt.axis('off') plt.xlim(0, 1) plt.ylim(0, 0.75**0.5) if border is True: plt.triplot(triangle, linewidth=1) fig = plt.figure() ax = fig.add_subplot(2,2,1) alpha=[3,3,3] r_diri = np.random.dirichlet(alpha, size=1000) ax.set_title("alpha={0}".format(alpha)) plot_points(r_diri) ax = fig.add_subplot(2,2,2) alpha=[0.3,0.3,0.3] r_diri = np.random.dirichlet([0.3,0.3,0.3], size=1000) ax.set_title("alpha={0}".format(alpha)) plot_points(r_diri) ax = fig.add_subplot(2,2,3) alpha=[3,0.3,0.3] r_diri = np.random.dirichlet([3,0.3,0.3], size=1000) ax.set_title("alpha={0}".format(alpha)) plot_points(r_diri) ax = fig.add_subplot(2,2,4) alpha = [1,1,1] r_diri = np.random.dirichlet([1,1,1], size=1000) ax.set_title("alpha={0}".format(alpha)) plot_points(r_diri) plt.show()

ガンマ分布
ガンマ分布は離散値を取る分布です。numpyの場合はshapeとscaleの2つのパラメータをとるのですが、scaleは教科書のの逆数になります。
というわけで
- 平均値は
- 標準偏差は
になります。ご注意下さい。
shape = 5 scale = 5 r_gamma = np.random.gamma(shape, scale, size=1000) g = sns.distplot(r_gamma, label="shape={0}, scale={1}".format(shape, scale), kde=False) shape = 5 scale = 20 r_gamma = np.random.gamma(shape, scale, size=1000) g = sns.distplot(r_gamma, label="shape={0}, scale={1}".format(shape, scale), kde=False) shape = 40 scale = 5 r_gamma = np.random.gamma(shape, scale, size=1000) g = sns.distplot(r_gamma, label="shape={0}, scale={1}".format(shape, scale), kde=False) g.legend() plt.show()

2変量正規分布
平均と分散にどう2変量が関わるか示す相関係数を入れ込みます。共分散行列の書き方はもっと綺麗なものがありそうですが。。
import seaborn as sns import matplotlib.pyploy as plt import numpy as np mu1 = 5 mu2 = 5 sigma1 = 10 sigma2 = 40 rho = 0.6 mu = np.array([mu1, mu2]) cov = np.array([[sigma1**2, sigma1*sigma2*rho], [sigma1*sigma2*rho, sigma2**2]]) r_mnorm = np.random.multivariate_normal(mu, cov, size=1000) g = sns.jointplot(r_mnorm[:, 0], r_mnorm[:, 1], kind="kde", n_levels=50) plt.show(g)

コーシー分布
numpyではパラメータを選べない標準コーシー分布しか使えません。そのためscipyを利用しました。
なおここまでのnumpyで生成した他の分布も、rvs(random variates sampling?)メソッドを使えば同様に得ることが出来ます。 ただnumpyとscipyで仕様が異なり、numpyの正規分布を由来とする乱数はnumpy.random.normalなのに、scipyはscipy.stats.normだったりします(解せぬ)。
コーシー分布は面白い分布ですね。パラメータがあるものの、平均値や分散をそこから推定できない。しかもめちゃめちゃ変なところに外れ値みたいに値が登場する性質を持っています。
import seaborn as sns import matplotlib.pyploy as plt from scipy.stats import cauchy r_cauchy = cauchy.rvs(0, 1, 100) sns.distplot(r_cauchy) plt.show()

練習問題(3)
のような分布になります。
これは以前に触れた部分ですね。
正規分布の線形結合で表される分布は、記事内のpdfを読めば全部同じように表されることが分かります。
y1 = np.random.normal(50, 20, 2000) y2 = np.random.normal(20, 15, 2000) diff = y1 - y2 sns.distplot(diff) plt.show()

練習問題(4)
何か自分でテキストに載っていない分布を対象にすること。という問題でした。
今回は負の二項分布を取り扱ってみます。生物学の分野では、遺伝子発現量(RNA-seq or cDNA-seq)の推定モデル(DESeqやedgeR)でよく使われている分布です。負の二項分布はポアソン分布とガンマ分布の混合モデルです。ホクソエムさんの分かりやすい解説があるので、そちらをご覧ください。ポアソン分布のが確率的に変動することで、過分散に対応できるようになっています。
負の二項分布の解説は英語版Wikipediaを見るのが一番いいと思います。以下そこからのざっくりとした抽出です。
「負の」の意味
負の二項分布は定義4パターン存在します。4つはそれぞれ
- 成功率pの試行をr回失敗(成功)するまでに、成功(失敗)した回数kの分布
- 成功率pの試行をr回失敗した時の、試行回数nの分布
- 成功率pの試行をk回成功した時の、失敗回数rの分布
- 成功率pの試行をk回成功した時の、試行回数nの分布
となります。4つのパターンがありますが、一番良く使われるのは1つ目のものです。
1つ目のパターンを式で表すと、

となります。このパターンは試行が成功でも失敗でも式に大きな変化がなく、逆で紹介されている場合もあります。その時は逆側の事象が起こる確率が1-pになるように対応が変化するだけです。ところで、成功回数と失敗回数の和が試行回数です。つまりとなります。これを使って整理すると

となりました。ところで普通の二項分布の式は次のようになります。

つまり負の二項分布だと二項分布と比べてトライアルの数が-1されていることになります。
平均と分散
負の二項分布の平均と分散は次のようになります。
詳しい導出についてはこちらを参考にするのが適切だと思いました。日本語版Wikipediaの記事は、どこか間違っているような…。
実数への拡張
負の二項分布のパラメータは正の実数へ拡張することが出来ます。「rが実数の『成功率pの試行をr回失敗(成功)するまでに、成功(失敗)した回数kの分布』ってなんやねん」と僕は思いました。おそらくここで肝心なのは、
を正の実数に拡張しても元々の定義へ矛盾せずに一般化できるということではないかと思います。この時ガンマ関数
を利用して、
のように書くことが出来ます。
平均と形状パラメータによる分布の指定
負の二項分布のパラメータを指定する時にではなく、平均パラメータ
と形状パラメータ
を使って記述されることがあります。形状パラメータとは分散をスッキリさせるために
の逆数を取って
としたものです。一応形状パラメータと書きましたが、他にも呼び方が色々あり、
- shape parameter
- dispersion parameter
- clustering coefficient
- heterogeneity
- aggregation
のようにとりとめがありません。文字の使い方も適当らしく、論文によってはが使われているものもありました。
分布の可視化
可視化をする時にbinsを自分で指定しています。何も指定しないと最適なbinsをseabornが決めてくれるのですが、範囲が小数で分割された場合には離散値である負の二項分布の値がダブルカウントされるのか、物凄く変な分布になってしまいます。気をつけて下さい。
import numpy as np from scipy.stats import nbinom import seaborn as sns import matplotlib.pyplot as plt # r_nb = np.random.negative_binomial(10, 0.3, 10000) r_nb = nbinom.rvs(10, 0.3, size=10000) sns.distplot(r_nb, kde=False, bins=np.arange(0,100,5)) plt.show()

余談ですがedgeRの論文は事実上、RNA-seqの解析に負の二項分布をあてはめだだけの論文です。2ページしかありません。そのわりに1000回を超えれば場外ホームランとなる世界で、2500回も引用されている(Scopus調べ)ので、なんとも夢があります。
StanとPythonでベイズ統計モデリング その2 Chapter5
アヒル本(StanとRでベイズ統計モデリング)のChapter5にPythonで取り組んでいきます。 練習問題を解いて、本文中に書かれてるグラフをPythonで描いてみます。
なおChapter1~3は導入だったのと、Chapter4は練習問題の内容が「はじめての統計データ分析」と被っていたのでパスします。
Chapter5 基礎的な回帰とモデルのチェック
- 重回帰
- 複数の説明変数を用いた回帰のこと
- 重回帰も結局は正規分布を仮定している
- 目的
- 説明変数からの応答変数の予想、及び説明変数の寄与率
- 分布
- 複数の説明変数ならScatterplot matrixを利用すると良い
- MCMCの設定について
- 結果の解釈 : モデルの改善に活かす!
- 複数の説明変数を用いた回帰のこと
- (二項)ロジスティック回帰
- ロジスティック関数
をつかった方法
- 応答変数が0から1の間にある場合、説明変数に適用することで値の範囲を変換することが出来る
- 2項分布モデル
- ベルヌーイ分布モデル
- 応答変数がbool値であるような時にはベルヌーイ分布を利用すれば良い
- 生成量
- オッズ
- 二項ロジスティック回帰モデルにおいて、
であり、
であるならば
と変換することが出来る
はオッズと呼ばれる
- その事象が起こるサンプリング確率の比率を示す
- 二項ロジスティック回帰モデルにおいて、
- オッズ
- 可視化
- ロジスティック関数
- ポアソン回帰
- カウントデータに対して予測を行う時に用いる
でカウントデータYが決定されるものとして、
は説明変数の線形結合を0以上に変換したものを使う(
がよく使われる)
- パフォーマンスの理由より、
が十分に大きいなら、正規分布で近似して良い
- カウントデータに対して予測を行う時に用いる
練習問題
Scatterplot matrixについて
Pythonの実装はpandasマスターのid:sinhrksさんの記事を見るのが良いと思います。
個人的な使った感想は次の通りです。
- 離散値の可視化が上手く行かないことがある(6離散値指定したのに、4つしか表示されないことがある)?
- たぶんこれはseabornのPairPlotのバグです……
- 離散値/連続値判定の初期値が大きすぎると上手くいかないので、離散値の判定閾値を4, 5辺りにいじって使いました。
- 離散値の場合は対角のカウントはcountplotを使ったほうが良い?
- 他の下半分、上半分のグラフと軸を合わせやすくするためです。
- どうせseabornはPairGridで使うことになりますし…
そういうわけで改造させて頂いたバージョンがこちらです。残念ながら1つ目のバグは取れていません。そもそも僕の環境に特有なのかもしれませんし、諦めました。 上三角行列の部分はid:statmodelingさんの記事のまんまです。
#!/usr/bin/env python # vim:fileencoding=utf-8 # Created: 2017-06-06 # Original scripts: http://sinhrks.hatenablog.com/entry/2016/11/01/075527 import matplotlib.pyplot as plt import numpy as np import pandas as pd import seaborn as sns from matplotlib.patches import Ellipse class Dispatcher(object): def __init__(self, fontsize=20, alpha=0.6, cmap='RdBu', threshold=5): self.fontsize = fontsize self.alpha = alpha self.cmap = plt.get_cmap(cmap) # 離散値 / 連続値とみなす閾値 self.threshold = threshold def comb(self, x_series, y_series, label=None, color=None): """ 下三角部分のプロット """ x_nunique = x_series.nunique() y_nunique = y_series.nunique() if x_nunique < self.threshold and y_nunique < self.threshold: # 離散値 x 離散値のプロット return self._dd_plot(x_series, y_series, label=label, color=color) elif x_nunique < self.threshold or y_nunique < self.threshold: # 離散値 x 連続値のプロット return self._dc_plot(x_series, y_series, label=label, color=color) else: # 連続値 x 連続値のプロット return plt.scatter(x_series, y_series, label=label, color=color) def _dd_plot(self, x_series, y_series, label=None, color=None): """ 離散値 x 離散値のプロット """ # x, y 各組み合わせの個数を集計 total = y_series.groupby([x_series, y_series]).count() # x, y軸をプロットする位置を取得 xloc = total.index.labels[0] yloc = total.index.labels[1] values = total.values ax = plt.gca() for xp, yp, vp in zip(xloc, yloc, values): ax.annotate(vp, (xp, yp), fontsize=self.fontsize, ha='center', va='center') # 組み合わせの個数を散布図としてプロット size = values / (values.max() * 1.1) * 100 * 20 ax.scatter(xloc, yloc, s=size, label=label, color=color) ax.set_ylim(yloc[0] - 0.5, yloc[-1] + 0.5) def _dc_plot(self, x_series, y_series, label=None, color=None): """ 離散値 x 連続値のプロット """ if y_series.nunique() < x_series.nunique(): # y軸が離散値の場合は、x, yを入替 # 水平方向に箱ひげ図をプロット orient = "h" else: orient = "v" ax = sns.boxplot(x_series, y_series, orient=orient, color=color) ax = sns.swarmplot(x_series, y_series, orient=orient, color=color) def diag(self, x_series, label=None, color=None): """ 対角部分のプロット """ x_nunique = x_series.nunique() if x_nunique < self.threshold: ax = sns.countplot(x_series, color=color) else: ax = sns.distplot(x_series, kde=False, color=color) ax.yaxis.set_visible(False) def ellipse(self, x_series, y_series, label=None, color=None): """ 上三角部分のプロット """ # 相関係数を楕円としてプロット r = x_series.corr(y_series) c = self.cmap(0.5 * (r + 1)) ax = plt.gca() ax.axis('off') artist = Ellipse(xy=[.5, .5], width=np.sqrt(1+r), height=np.sqrt(1-r), angle=45, facecolor=c, edgecolor='none', transform=ax.transAxes) ax.add_artist(artist) ax.text(.5, .5, '{:.0f}'.format(r*100), fontsize=28, horizontalalignment='center', verticalalignment='center', transform=ax.transAxes) d = Dispatcher() # データを作る x = np.random.normal(100, 5, 30) y = np.random.choice([1,2,3], 30, p=[0.1,0.2,0.7]) z = np.random.choice([1,2,3,4], 30, p=[0.2,0.2,0.2,0.4]) w = np.random.normal(4, 0.1, 30) df = pd.DataFrame(np.array([x,y,z,w]).T, columns=["x", "y", "z", "w"]) # 可視化する g = sns.PairGrid(df, diag_sharey=False) g.map_diag(d.diag) g.map_lower(d.comb) g.map_upper(d.ellipse) plt.show()
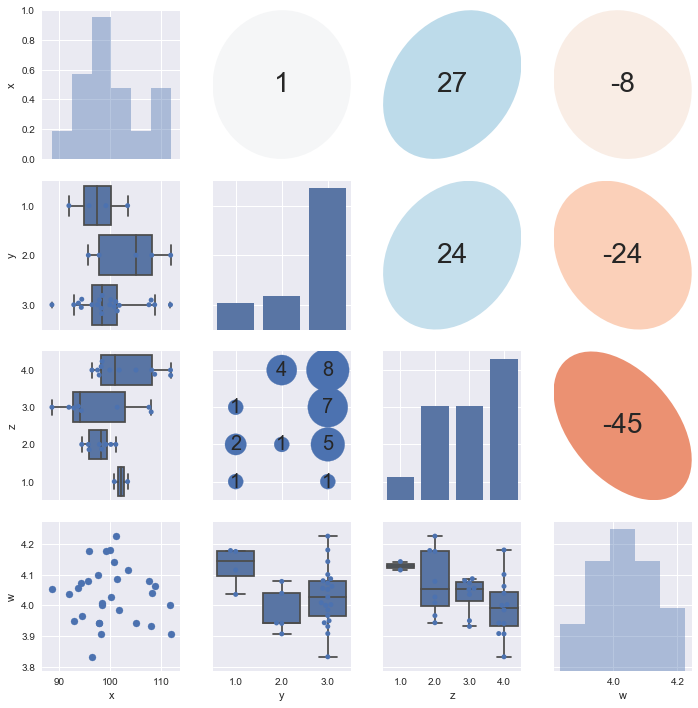
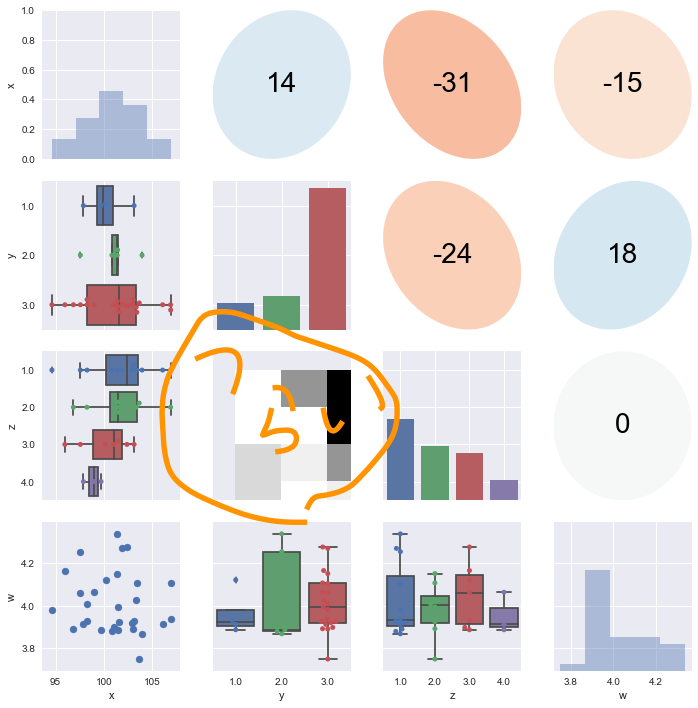
これとは別に、勉強として自分でも実装してみましたが、離散×離散の可視化が難しいところです。僕はヒートマップを使おうかと思ったのですが、軸を表示しようとする関係でズレます。つらい。バブルプロットを使うのがテキスト通りの適切なアイディアです。 一応載せておきます。
import matplotlib.pyplot as plt import numpy as np import pandas as pd import seaborn as sns from matplotlib.patches import Ellipse from scipy import stats def value_type(v): if v.unique().size < 5: v_type = "discrete" else: v_type = "continuous" return v_type def correlation(x, y, **kws): r, _ = stats.spearmanr(x, y) ax = plt.gca() ax.axis('off') ellcolor = plt.cm.RdBu(0.5*(r+1)) txtcolor = 'black' if np.absolute(r) < 0.5 else 'white' artist = Ellipse(xy=[.5, .5], width=np.sqrt(1+r), height=np.sqrt(1-r), angle=45, facecolor=ellcolor, edgecolor='none', transform=ax.transAxes) ax.add_artist(artist) ax.text(.5, .5, '{:.0f}'.format(r*100), color=txtcolor, fontsize=28, horizontalalignment='center', verticalalignment='center', transform=ax.transAxes) def diagonal(x, **kws): ax = plt.gca() x_type = value_type(x) if x_type == "discrete": sns.countplot(x) else: sns.distplot(x, kde=False) def distribution(x, y, **kws): ax = plt.gca() x_type = value_type(x) y_type = value_type(y) if x_type == "discrete": if y_type == "discrete": df = pd.DataFrame([x, y]).T df.columns=[x.name, y.name] pdf = df.pivot_table(index=x.name, columns=y.name, aggfunc=lambda x: len(x)).fillna(0).T plt.pcolor(pdf) else: sns.boxplot(x, y) sns.swarmplot(x, y) else: if y_type == "discrete": sns.boxplot(x, y, orient="h") sns.swarmplot(x, y, orient="h") else: plt.scatter(x, y) def scatterplot_matrix(df): g = sns.PairGrid(df) g.map_upper(correlation) g.map_diag(diagonal) g.map_lower(distribution) return g x = np.random.normal(100, 5, 30) y = np.random.choice([1,2,3], 30, p=[0.1,0.2,0.7]) z = np.random.choice([1,2,3,4], 30, p=[0.4,0.3,0.2,0.1]) w = np.random.normal(4, 0.1, 30) df = pd.DataFrame(np.array([x,y,z,w]).T, columns=["x", "y", "z", "w"]) g = scatterplot_matrix(df) plt.show(g)
練習問題(1)
まぁほとんど弄る部分もありません。 いつも通りPyStanを回すだけです。
import pystan import pandas as pd import os import pickle from IPython.core.display import display stan_file = os.path.join("RStanBook", "chap05", "model", "model5-3.stan") model = pystan.StanModel(stan_file) df = pd.read_csv("./RStanBook/chap05/input/data-attendance-1.txt") N = len(df) A = df["A"].values Score = df["Score"].values / 200 Y = df["Y"].values stan_data = { "N": N, "A": A, "Score": Score, "Y": Y } fit = model.sampling( data=stan_data, iter=2100, chains=4, warmup=100, seed=1234, ) summary = fit.summary() summary_df = pd.DataFrame( summary["summary"], index =summary["summary_rownames"], columns=summary["summary_colnames"] ) display(summary_df) mu = fit.extract()["mu"] diff = np.apply_along_axis(lambda x: Y - x, axis=1, arr=mu)
モデルはgithubで公開されているものをそのまま使いました。
練習問題(2)
Stanの内部の生成量を出すところで残差を計算します。 個人的には汚い部分を全部Stanの中に隠せるのでこっちのほうが好きです。
import pystan import pandas as pd import os import pickle from IPython.core.display import display stan_file = os.path.join("RStanBook", "chap05", "model", "model5-3_kai.stan") stan_file_c = os.path.join("RStanBook", "chap05", "model", "model5-3_kai.pkl") # コンパイルチェック if os.path.exists(stan_file_c) == False: # コンパイル済みのバイナリが無かったらstanファイルからコンパイル model = pystan.StanModel(stan_file) with open(stan_file_c, "wb") as f: pickle.dump(model, f) else: # バイナリがあったらそれを利用 with open(stan_file_c, "rb") as f: model = pickle.load(f) df = pd.read_csv("./RStanBook/chap05/input/data-attendance-1.txt") N = len(df) A = df["A"].values Score = df["Score"].values / 200 Y = df["Y"].values stan_data = { "N": N, "A": A, "Score": Score, "Y": Y } fit = model.sampling( data=stan_data, iter=2100, chains=4, warmup=100, seed=1234, ) summary = fit.summary() summary_df = pd.DataFrame( summary["summary"], index =summary["summary_rownames"], columns=summary["summary_colnames"] ) display(summary_df)
モデルは次の通りです。
data {
int N;
int<lower=0, upper=1> A[N];
real<lower=0, upper=1> Score[N];
real<lower=0, upper=1> Y[N];
}
parameters {
real b1;
real b2;
real b3;
real<lower=0> sigma;
}
transformed parameters {
real mu[N];
for (n in 1:N)
mu[n] = b1 + b2*A[n] + b3*Score[n];
}
model {
for (n in 1:N)
Y[n] ~ normal(mu[n], sigma);
}
generated quantities {
real y_pred[N];
real diff[N] ;
for (n in 1:N){
y_pred[n] = normal_rng(mu[n], sigma) ;
diff[n] = Y[n] - mu[n] ;
}
}
つづいてpythonでグラフを描いて、目視で比較をします。
まず実測値とモデルによる予測値を比較してみます。
テキストでは予測分布を使っていますが、試しにMCMCで推定されたパラメータ分布の中央値を使って値を予測してみます。この目的は、後述する事後予測分布との比較です。これは「はじめての統計データ分析」で言うところの、条件付き予測分布を使った手法です。こちらの手法だとStanファイル中で予測をしなくていい代わりに、エラーバーをつけるのがめんどくさいです。何故ならYを導出するためのにも
にも分布があるので、その両方を考慮する必要があります。今回はパスさせて下さい。
import numpy as np import seaborn as sns import matplotlib.pyplot as plt # Aの値でindexを分類する A0_samples = df[df["A"] == 0].index A1_samples = df[df["A"] == 1].index # summaryのtableからmuの中央値とsigmaを取得する summary_df_A0 = summary_df.loc[["mu[{0}]".format(i) for i in A0_samples], :] summary_df_A1 = summary_df.loc[["mu[{0}]".format(i) for i in A1_samples], :] map_A0 = summary_df_A0["50%"] map_A1 = summary_df_A1["50%"] sigma = summary_df.loc["sigma", "50%"] # パラメータを使って条件付き予測分布を推定する Y_A0 = Y[A0_samples] Y_A1 = Y[A1_samples] Y_A0A1 = np.hstack([Y_A0, Y_A1]) Y_ast_A0 = np.random.normal(map_A0, sigma) Y_ast_A1 = np.random.normal(map_A1, sigma) Y_ast_A0A1 = np.hstack([Y_ast_A0, Y_ast_A1]) # グラフのために相関係数を導出する r = np.corrcoef(Y_A0A1, Y_ast_A0A1)[0][1] # 可視化 fig, ax = plt.subplots() ax.plot(Y_ast_A0, Y_A0, "o", label="A=0") ax.plot(Y_ast_A1, Y_A1, "^", label="A=1") ax.plot([0, 0.5], [0, 0.5], "k-") ax.legend() # 体裁を整える ax.set_aspect('equal') ax.set_xlabel("Predicted value(by EAP)") ax.set_ylabel("Observed value") ax.set_title("r={0}".format(r)) plt.show()
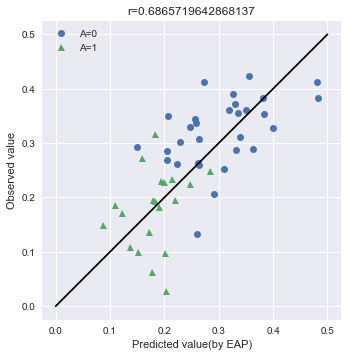
Aについてバラけているわけではないですが、あまり精度が良く無さそうです。モデルの精度が良くないのでしょうか(後述しますが、これは違います)?
続いて、MCMCサンプリングの中でyの予測値を推定します。これは「はじめての統計データ分析」で言うところの、事後予測分布を使った手法です。こちらのほうが分布全体を考慮して行っているので、正確な予測値を計算することが出来ます。エラーバーについてもMCMCの分布を使えばいいので簡単です。今回は教科書の80%区間ではなく、デフォルトで計算される75%区間を利用しています。
# Aの値でindexを分類する A0_samples = df[df["A"] == 0].index A1_samples = df[df["A"] == 1].index Y_A0 = Y[A0_samples] Y_A1 = Y[A1_samples] Y_A0A1 = np.hstack([Y_A0, Y_A1]) # プロット点の計算 summary_df_A0 = summary_df.loc[["y_pred[{0}]".format(i) for i in A0_samples], :] summary_df_A1 = summary_df.loc[["y_pred[{0}]".format(i) for i in A1_samples], :] Y_pred_A0 = summary_df_A0["50%"] Y_pred_A1 = summary_df_A1["50%"] Y_pred = np.hstack([Y_pred_A0, Y_pred_A1]) # 誤差の計算 Y_pred_A0_err_down = (Y_pred_A0 - Y_pred_summary_A0["25%"]).values Y_pred_A1_err_down = (Y_pred_A1 - Y_pred_summary_A1["25%"]).values Y_pred_A0_err_up = (Y_pred_summary_A0["75%"] - Y_pred_A0).values Y_pred_A1_err_up = (Y_pred_summary_A1["75%"] - Y_pred_A1).values Y_pred_A0_err = np.vstack([Y_pred_A0_err_down, Y_pred_A0_err_up]) Y_pred_A1_err = np.vstack([Y_pred_A1_err_down, Y_pred_A1_err_up]) # 相関係数の計算 r = np.corrcoef(Y_A0A1, Y_pred)[0][1] # 可視化 fig, ax = plt.subplots() ax.errorbar(Y_pred_A0, Y_A0, yerr=Y_pred_A0_err, fmt="o", label="A=0") ax.errorbar(Y_pred_A1, Y_A1, yerr=Y_pred_A1_err, fmt="^", label="A=1") # y=xの線を書く ax.plot([0, 0.5], [0, 0.5], "k-") # 凡例をつける ax.legend() # 体裁を整える ax.set_aspect('equal') ax.set_xlabel("Predicted value") ax.set_ylabel("Observed value") ax.set_title("r={0}".format(r)) plt.show()
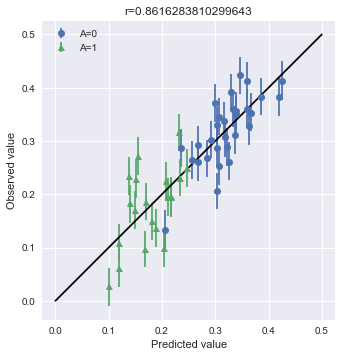
結果がわりと異なります。相関係数に如実に現れました。条件付き予測分布ではダメかと思ったモデルですが、こちらでは問題がなさげなことが推定されます。以上のことから、条件付き予測分布はオンラインで利用できるなどの利点がありますが、その分結果の浮動性があることを頭に入れたほうが良いと思います。とはいえ、どちらの方法でもAに関する偏ったバラつきが無いことはわかりますので、何を見たいかの使い分けが結局のところ肝心です。
最後に実測値とパラメータの誤差を調べます。
summary_df_diff = summary_df.loc[["diff[{0}]".format(i) for i in range(0, 50)], :] fig, ax = plt.subplots() sns.distplot(summary_df_diff["50%"], bins=np.arange(-0.15, 0.15, 0.03), ax=ax) ax.set_xlabel("Epsilon EAP") ax.set_ylabel("Count") plt.show()
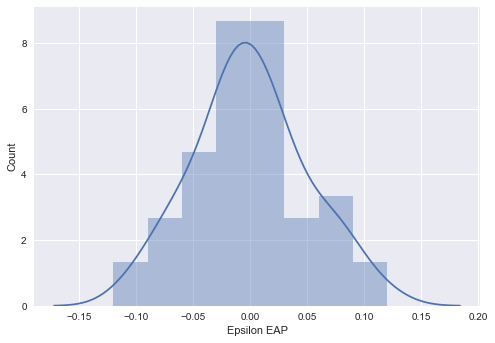
教科書の結果から分かっていましたが、きちんとあてはめた正規分布に従っています。
練習問題(3)
離散値と離散値の集計です。pandasのpivot_tableメソッドを使えばいいのですが、芸がないのでヒートマップで可視化しておきます。AとYの各組み合わせで足していけばいいので、ラムダ式を読み出してlenでカウントしました。
import pandas as pd import seaborn as sns import matplotlib.pyplot as plt df = pd.read_csv("./RStanBook/chap05/input/data-attendance-3.txt") pdf = df[["A", "Y"]].pivot_table(index="A", columns="Y", aggfunc=lambda x:len(x)) g = sns.heatmap(pdf, annot=True) plt.show(g)
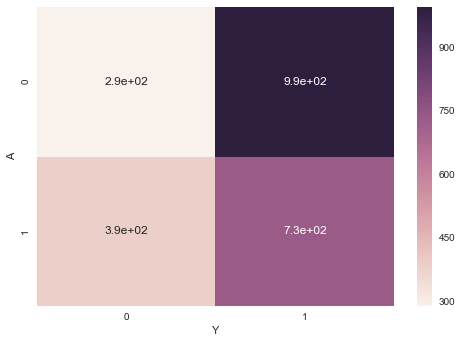
練習問題(4)
晴れがもたらす影響を基準値として、曇と雨が持つ影響についてもMCMCから推定してみます。これだけだと芸が無いので、WAICによるモデルのフィッティングについても評価してみます。
import pystan import pandas as pd import os import pickle import numpy as np from IPython.core.display import display from logging import getLogger, Formatter, StreamHandler, DEBUG # printではなくloggerを使う def get_logger(): logger = getLogger(__name__) logger.setLevel(DEBUG) log_fmt = '%(asctime)s : %(name)s : %(levelname)s : %(message)s' formatter = Formatter(log_fmt) stream_handler = StreamHandler() stream_handler.setLevel(DEBUG) stream_handler.setFormatter(formatter) logger.addHandler(stream_handler) return logger logger = get_logger() stan_file = os.path.join("RStanBook", "chap05", "model", "model5-5_kai.stan") model = pystan.StanModel(stan_file) trans = {"A": 1, "B": 2, "C": 3} df = pd.read_csv("./RStanBook/chap05/input/data-attendance-3.txt") I = len(df) A = df["A"].values Score = df["Score"].values / 200 W = df["Weather"].values W = np.array([trans[w] for w in W]) Y = df["Y"].values stan_data = { "I": I, "A": A, "Score": Score, "W": W, "Y": Y } fit = model.sampling( data=stan_data, iter=1200, chains=4, warmup=200, seed=1234, ) summary = fit.summary() summary_df = pd.DataFrame( summary["summary"], index =summary["summary_rownames"], columns=summary["summary_colnames"] ) display(summary_df) log_lik = fit.extract("log_lik")["log_lik"] waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0)) logger.info("WAICの値は{0}です".format(waic))
モデルは以下の通りです。
data {
int I;
int<lower=0, upper=1> A[I];
real<lower=0, upper=1> Score[I];
int<lower=1, upper=3> W[I];
int<lower=0, upper=1> Y[I];
}
parameters {
real b[3];
real b_w[2] ;
}
transformed parameters {
real q[I];
real beta_w[3] ;
beta_w[1] = 0 ;
beta_w[2] = b_w[1] ;
beta_w[3] = b_w[2] ;
for (i in 1:I)
q[i] = inv_logit(b[1] + b[2]*A[i] + b[3]*Score[i] + beta_w[W[i]]);
}
model {
for (i in 1:I)
Y[i] ~ bernoulli(q[i]);
}
generated quantities {
real log_lik[I] ;
for (i in 1:I){
log_lik[i] = bernoulli_lpmf(Y[i]|q[i]) ;
}
}
さて経験的にパラメータを決定した時と比べて、WAICはどうなったでしょうか。
- 経験的に決めた場合 : WAIC=2769
- MCMCからサンプリングした場合: WAIC=2764
ちょっとだけ、MCMCからサンプリングした場合のほうが値が良くなります。EAPを見てモデルを書いてみると
- 経験的に決めた場合
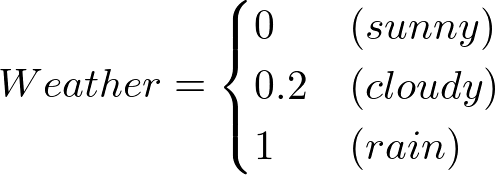
- MCMCからサンプリングした場合
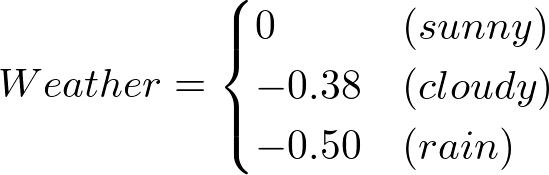
となります。Weatherの部分も係数の-0.45を考慮すると、そこまで変わりません。僕は経験的に得られた0:0.2:1が結構いいものであったと解釈しました。
つづいてpythonでグラフを描いて、目視で結果を比較します。 テキストに描いてあるように、ロジスティック回帰の観測値は0/1になるので、可視化が難しいところです。qの値が、データからどのように変化するかを調べます。テキストの例では連続値のScoreと離散値のAが可視化されています。
まずScoreの影響を可視化します。
- 推測したqの中央値をデータと対応付けるために、indexを振り直します。
- カラムを複数使ったデータの選択なので、queryメソッドで取得します。
- qの中央値の線をきれいに書くためにソートを挟んでいます
# qの値を取得 summary_df_q = summary_df.loc[["q[{0}]".format(i) for i in range(0, I)], :] q_range = summary_df_q[["25%", "50%", "75%"]] q_range.index = range(len(q_eap)) # データと結合(順番はこの時点では保存されている) df_concat = pd.concat([df, q_range], axis=1) # 一部のデータだけ選択 df_filtered = df_concat.query("A == 0 & Weather == 'A'") df_filtered = df_filtered.sort_values("Score") # Yの点の位置をそれぞれちょっとずらす y_randomized = [y + np.random.normal(0, 0.05) for y in df_filtered["Y"]] # 可視化 fig, ax = plt.subplots() ax.plot(df_concat_filtered["Score"], df_concat_filtered["50%"], "k-") ax.fill_between(df_concat_filtered["Score"], df_concat_filtered["25%"], df_concat_filtered["75%"], facecolor="pink") ax.plot(df_concat_filtered["Score"], y_randomized, "o", label="Y") ax.legend() ax.set_xlabel("Score") ax.set_ylabel("q") plt.show()
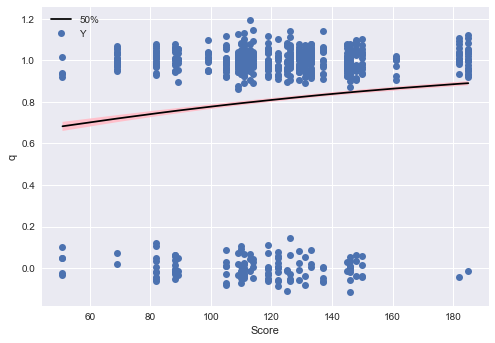
次にAに対するプロットを書きましょう。matplotlibで頑張るのが面倒だったのでseabornで書きました(なんかごめんなさい)。適材適所です!
# qの値を取得 summary_df_q = summary_df.loc[["q[{0}]".format(i) for i in range(0, I)], :] q_range = summary_df_q[["25%", "50%", "75%"]] q_range.index = range(len(q_eap)) # データと結合(順番はこの時点では保存されている) df_concat = pd.concat([df, q_range], axis=1) # 可視化 fig, ax = plt.subplots() sns.violinplot(data=df_concat, x="50%", y="Y", ax=ax, orient="h") sns.swarmplot(data=df_concat, x="50%", y="Y", hue="A", ax=ax, orient="h", palette=sns.color_palette("Set2", n_colors=2)) ax.set_xlabel("q") ax.set_ylabel("Y") plt.show()
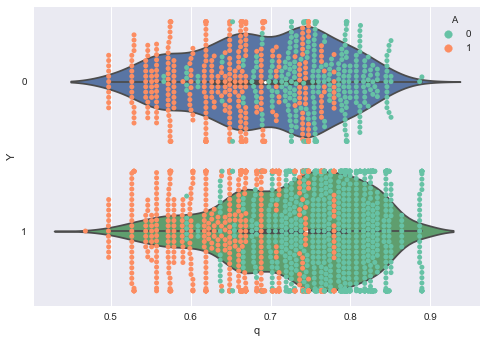
最後にROCカーブを書いて、AUCを計算します。これについてはScikit-learnを使うのが簡単でしょう。
from sklearn.metrics import roc_curve, roc_auc_score summary_df_q = summary_df.loc[["q[{0}]".format(i) for i in range(0, I)], :] q = summary_df_q["50%"] # AUCの計算 auc = roc_auc_score(Y, q) # ROCカーブの座標を計算 false_positive_rate, true_positive_rate, _ = roc_curve(Y, q) fig, ax = plt.subplots() ax.plot(false_positive_rate, true_positive_rate) # y=xを描く ax.plot([0, 1], [0, 1], "k-") # 体裁を整える ax.set_aspect('equal') ax.set_xlabel("False Positive Rate") ax.set_ylabel("True Positive Rate") ax.set_title("AUC = {0}".format(auc)) plt.show()
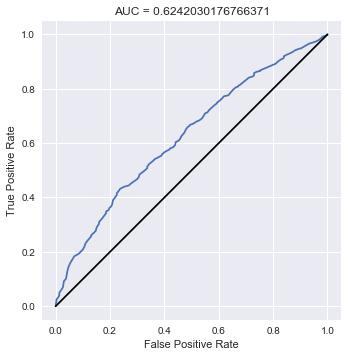
練習問題(5)
import matplotlib.pyplot as plt import os import pandas as pd import pickle import pystan from IPython.core.display import display stan_file = os.path.join("RStanBook", "chap05", "model", "model5-6b.stan") model = pystan.StanModel(stan_file) trans = {"A": 1, "B": 2, "C": 3} df = pd.read_csv("./RStanBook/chap05/input/data-attendance-2.txt") N = len(df) A = df["A"].values Score = df["Score"].values / 200 M = df["M"].values stan_data = { "N": N, "A": A, "Score": Score, "M": M } fit = model.sampling( data=stan_data, iter=2100, chains=4, warmup=100, seed=1234, ) summary = fit.summary() summary_df = pd.DataFrame( summary["summary"], index =summary["summary_rownames"], columns=summary["summary_colnames"] ) display(summary_df) Mpred = np.median(fit.extract()["m_pred"], axis=0) fig = plt.figure(figsize=(5,5)) ax = fig.gca() ax.plot(Mpred, M, "o") ax.plot(range(0, 100), range(0, 100)) ax.set_xlim(0, 100) ax.set_ylim(0, 100) ax.set_xlabel("Predicted") ax.set_ylabel("Measured") plt.show(ax)
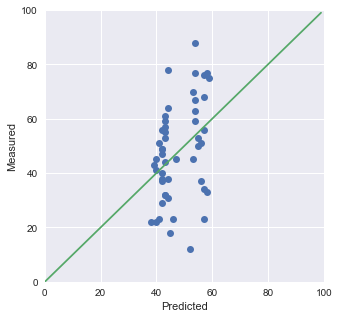
全然y=xに乗っていません…(合っているのかめちゃめちゃ不安です)。
この結果からは、予測が出来ていないことが分かります。説明変数が足りてないということですね。よくあることですが、計測が終わった後にこうなった場合は、新しくメタデータを足して計測し直しになるわけです。つらい。
練習問題(6)
練習問題(5)と(6)はちょっと気をつける必要があります。アヒル本だけ読んだ時には「処理」の違いというものが
- 両方に別々の処理を施した
- 片方には処理を施して、片方には何もしなかった
という2つのパターンのどちらなのか分かりません。前者の場合にはベースラインとなるものに別々の効果が生じることを予測できますが、後者の場合には一方だけはベースラインのままで、もう一方に何か効果が現れることになります。
データの元となった緑本を読んでみると、後者であることが分かります。前者を踏まえてモデルを作ってみると、MCMCが収束せずに頭を悩まるハメになります(僕は緑本を読んだはずなのに忘れていて喘ぎました)。
前者の場合でも相対的な効果を踏まえると同じじゃないの?と思っていたのですが収束しないですし、そうではないんですね。ちょっとした発見でした。これはChapter 10の一番初めに出てくる識別可能性の問題に引っかかるからですね。
import pystan import pandas as pd import os import pickle from IPython.core.display import display stan_file = os.path.join("RStanBook", "chap05", "model", "practice6.stan") model = pystan.StanModel(stan_file) trans = {"C": 0, "T": 1} df = pd.read_csv("./RStanBook/chap05/input/data3a.csv") N = len(df) X = df["x"].values F = df["f"].values F = np.array([trans[x] for x in F]) Y = df["y"].values stan_data = { "N": N, "X": X, "F": F, "Y": Y } fit = model.sampling( data=stan_data, seed=1234, ) summary = fit.summary() summary_df = pd.DataFrame( summary["summary"], index =summary["summary_rownames"], columns=summary["summary_colnames"] ) display(summary_df)
モデルは次です。
data {
int N;
real<lower=0> X[N];
int<lower=0, upper=1> F[N];
int<lower=0> Y[N];
}
parameters {
real b[3] ;
}
transformed parameters {
real<lower=0> lambda[N] ;
for (n in 1:N){
lambda[n] = exp(b[1] + b[2]*X[n] + b[3] * F[n]) ;
}
}
model {
for (n in 1:N){
Y[n] ~ poisson(lambda[n]);
}
}
練習問題(7)
(6)と同様に処理の効果にだけ気をつけて下さい。
import pystan import pandas as pd import os import pickle from IPython.core.display import display stan_file = os.path.join("RStanBook", "chap05", "model", "practice7.stan") model = pystan.StanModel(stan_file) trans = {"C": 0, "T": 1} df = pd.read_csv("./RStanBook/chap05/input/data4a.csv") I = len(df) N = df["N"].values X = df["x"].values F = df["f"].values F = np.array([trans[x] for x in F]) Y = df["y"].values stan_data = { "I": I, "N": N, "X": X, "F": F, "Y": Y } fit = model.sampling( data=stan_data, iter=1500, chains=4, warmup=500, seed=1234, ) summary = fit.summary() summary_df = pd.DataFrame( summary["summary"], index =summary["summary_rownames"], columns=summary["summary_colnames"] ) display(summary_df)
モデルは以下の通りです。
data {
int I;
int<lower=0> N[I];
real<lower=0> X[I];
int<lower=0, upper=1> F[I];
int<lower=0> Y[I];
}
parameters {
real b[3] ;
}
transformed parameters {
real<lower=0> q[I] ;
for (i in 1:I){
q[i] = inv_logit(b[1] + b[2]*X[i] + b[3]*F[i]) ;
}
}
model {
for (i in 1:I){
Y[i] ~ binomial(N[i], q[i]) ;
}
}
練習問題(4), (6)と(7)は初めに取り組んだ時に、モデルが全然収束しなくて足踏みしました。そういう時にはMCMCサンプリングが無理をして変な部分を探索しようとするのでめちゃめちゃ時間がかかります。なので待機時間で「失敗したな」と悟れたりするのはちょっと面白いところです。
StanとPythonでベイズ統計モデリング その1
StanとRでベイズ統計モデリング(通称アヒル本)をだいたい読みました。

StanとRでベイズ統計モデリング (Wonderful R)
- 作者: 松浦健太郎,石田基広
- 出版社/メーカー: 共立出版
- 発売日: 2016/10/25
- メディア: 単行本
- この商品を含むブログ (8件) を見る
本の紹介
既に様々な書評もありますし、方々から賛辞の声を挙げられている本です。僕としても非常に分かりやすく、使える本だと感じました。著者の松浦さんがウリを書いてくださっているので、まずそれを読むのが良いと思います。様々な方の書評も纏められています。
読んでみての感想を、良い点と改訂版に期待する点(笑)で書いてみたいと思います。
良い点
使えそうな分布が結構紹介されている
これは僕の専門分野が生命情報解析、著者の松浦さんの専門が医療統計で近いということもあるのですが、使えそうでありつつ統計入門レベルでは出てこない分布が実例を交えて紹介されています。例えば
- コーシー分布
- ゼロ過剰ポアソン分布
- ディリクレ分布
などです。ここらへんは数式だけではよく分かりませんし、とっつきづらいなところです。Stanと交えながら活用例を紹介してくださっているのは、イメージがつかみやすかったです。
階層モデル以外のベイジアンモデリングがふんだんに登場する
ベイジアンモデリングに挑戦してみよう、となると久保先生の通称緑本が第一の選択肢にあがります。僕も1年前に読みました。緑本はベイズ化する前の線形モデルから始まり、ベイズまで展開していくことで個人差が生じる過程をどう解析するのか非常に分かりやすく理解することが出来ます。

データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学)
- 作者: 久保拓弥
- 出版社/メーカー: 岩波書店
- 発売日: 2012/05/19
- メディア: 単行本
- 購入: 16人 クリック: 163回
- この商品を含むブログ (29件) を見る
ただ、場所差、個体差などのカテゴリーの違いで本が終わってしまうので「ベイズ化すればパラメータ推定が楽になるんだな」ぐらいの感覚におちついてしまいました。これは緑本が「ベイジアンモデル」の本ではなく、「モデリング」の本ということです。
アヒル本はベイズモデルに特化しており、階層モデル以外にも
- 状態空間モデル
- トピックモデル
- 打ち切りや外れ値への対応
が紹介されています。そのためベイジアンモデルの「あいまいさ自体をパラメータ化してしまう」という特徴が他にどのように活かせるのか理解しやすく、応用範囲の広さにも気が付くことが出来ました。
ベイズモデルの難しいところは、「パラメータを生み出す分布がある」という感覚の理解かと思っています。トピックモデルなんかは、これがないと数式がごっついので難解ですし。アヒル本はその感覚の養成に役立ちます。
有用なMCMCを使ったベイジアンモデルの参考書
Stanを使ったMCMCでのモデルフィッティングにおいて、想定される問題点の解決策が述べられています。
ここらへんはStanのマニュアルを読めば書いてある事象ではあります。ただ僕は読んでみても行間が抜けすぎていて、わからない部分が結構出てきました。 なんたって、600ページありますから…。
アヒル本はマニュアルの時には行間として省略されていたところを初学者向けに補足してくれているので、緑本を読んだ後ぐらいだったら殆ど詰まること無く読めました。現状だと確率的プログラミング言語としてEdwardがアツい印象がありますが、アヒル本はベイジアンモデルの内容を包括的に書いてくれているのでStanが使えなくなったから無駄になる知識ではなさそうです。
文法が新しい
Stanは2.10.0辺りで新しい文法が提案されたようです。そのためマニュアルを読んでも、本中で紹介されているtarget記法が出てこなかったりします。これは2.9.0を参考に和訳されている日本語マニュアルでも同様です。
アヒル本の文法は、2017-06-14時点では問題なく新しいバージョンで書かれています。なのでエラーメッセージにうんざりすることもありません。
改訂版に期待する点
紹介した分布を全部使って欲しい
6章で分布を幾つか紹介しています。先述したコーシー分布やディリクレ分布などはここで登場します。
ただ、ここでラプラス分布は紹介されるものの、本中ではそれっきりです。 ぜひこれらの分布についても実例を挙げて解説していただき、活かせる点を見せてくれたらいいなと思いました。きっと何かしらの有用性があると思いますので。
練習問題を面白くして欲しい
4章以降は各章の最後に練習問題が載っています。学習した内容を復習・確認することが出来ますし、答えについてもレポジトリに R + Stanの実装で配布されています。最高です。
ただあくまで確認が目的なのか、他の参考書のデータを引用していたり、本文中の内容の焼き直しだったりしたので退屈に感じました。練習問題に+alphaの内容を盛り込んで、更なる学習が出来ればよりモチベーションアップに繋がるのではないでしょうか。
書評はこんな感じです。 「はじめての統計データ分析」みたいに全部の問題をやったりはしませんが、Python + PyStanで幾つか解いてみたいと思います。タイトルはそういう意味です笑。
はじめての 統計データ分析 ―ベイズ的〈ポストp値時代〉の統計学― その7
その7です。今回は第6章の章末問題に取り組んでいきます。
6章 比率とクロス表の推測
内容
- 離散的な値をとるカウントデータの解析
カテゴリカル分布 : カテゴリカルな分類においてカウントとして得られるデータに対応する分布
- ベルヌイ試行
- 2項分布
- 多項分布
- 各事象が起こる確率が独立であることに注意
1つの2項分布の比率の推測:「きのこの山とたけのこの山の好きな人の比率」など
- 確率の事前分布は無情報であるので、
を仮定してMCMCを行う
- (二項分布を仮定した時の)事象が生起する確率
を推測することが可能
- 生成量
- オッズ :
- 2つの選択肢のどちらの確率が大きいかを測ることが出来る
- 仮説(基準とした確率値
に対して、pが下回る確率など; 即ち
)の検証
- オッズ :
- 確率の事前分布は無情報であるので、
1つの多項分布の比率の推測
- 同様に事前分布は無情報であるので、
番目のカテゴリの事前分布に
を仮定してMCMCを行う
- 全ての確率の和が1となるので、カテゴリが全部でn個なら n個目は
として決定される制限がある
- 全ての確率の和が1となるので、カテゴリが全部でn個なら n個目は
- 0からn番目の事象が独立してそれぞれ起こるのであれば、母数
が発生する確率は1つ1つの事象を
として
である
- 生成量
- カテゴリ間の比較(
など)
- 連言命題が正しい確率
- カテゴリ間の比較(
- 同様に事前分布は無情報であるので、
複数の2項分布の推測(独立したクロス表) : カテゴリが
個あって、それぞれで2つの観測結果が有った際にカテゴリ毎に or 全体でどのような差があるかを調べたい場合
- (独立した)2 × 2のクロス表
- 興味があるベルヌイ試行の確率だけ見る
- 男と女でベルヌイ試行の確率が異なる(独立した2項分布が2つある)と考える
- 生成量
- 比率の差:
, 性質の比:
- 性質の違いを考えることが出来る
- オッズ比 :
- 正反応は他方の反応の何倍生じやすいか、を表す
- 比率の差:
- g × 2 のクロス表
- 各f(theta)が得られる確率については何もわからないので、
の無情報事前分布を採用
- (独立した)2 × 2のクロス表
対応あるクロス表の推測(構造のある多項分布): 1つの標本から2回測定が有った場合
- 2 × 2のクロス表: 2つの事象の発生にどれだけ関係があるか?
- 2つの分布が独立ではなく、対応がある場合
- あるいはベルヌイ試行の興味が2つの分布の同時性にある場合
;
- 独立と連関
- 変数Aのカテゴリ
, 変数Bのカテゴリ
が独立である場合、
- ピアソン残差 :
- セルごとに計算され、独立性/連関性を示す
- 独立である場合0となる
- 正の値は独立の場合より観測されやすく、負の場合は独立の場合より観測されづらいことを示す
- クラメルの連関係数 :
;
はカテゴリの総数
- クロス表全体での連関の大きさを示す
- 小さいほど独立、大きいほど連関である
- 変数Aのカテゴリ
- 2 × 2のクロス表: 2つの事象の発生にどれだけ関係があるか?
章末問題
前準備
import os from logging import getLogger, Formatter, StreamHandler, DEBUG # printではなくloggerを使う def get_logger(): logger = getLogger(__name__) logger.setLevel(DEBUG) log_fmt = '%(asctime)s : %(name)s : %(levelname)s : %(message)s' formatter = Formatter(log_fmt) stream_handler = StreamHandler() stream_handler.setLevel(DEBUG) stream_handler.setFormatter(formatter) logger.addHandler(stream_handler) return logger logger = get_logger() # 出力用ディレクトリの用意 result_dir = os.path.join("result", "chapter6") if os.path.exists(result_dir) is False: os.makedirs(result_dir) logger.info("{0} is made".format(result_dir))
2項分布によるコインの予測
大学入試みたいに表と裏のでる確率が異なるイカサマコインではなく、普通の等確率なコインを想定しました。
from scipy.stats import binom prob = binom.pmf(3, 5, 0.5) logger.info("コインを5回投げて3回表が出る確率は{0}".format(prob))
多項分布によるじゃんけんの予測
from scipy.stats import multinomial prob = multinomial.pmf([2,2,1], 5, [0.3, 0.3, 0.4]) logger.info("グー:チョキ:パー = 2:2:1となる確率は{0}".format(prob))
MCMCによるベルヌイ試行の確率推定
Stanによる統計モデルの構築
import os import pystan import pickle # Stanのモデルを読み込んでコンパイルする stan_file = os.path.join("stan", "binomial.stan") stan_file_c = os.path.join("stan", "binomial.pkl") model = pystan.StanModel(file=stan_file) with open(stan_file_c, "wb") as f: pickle.dump(model, f) logger.info("Stan model is compiled to {0}".format(stan_file_c))
- binomial.stan
data {
int<lower=0> g ;
int<lower=0> x[g] ;
int<lower=0> n[g] ;
}
parameters {
real<lower=0, upper=1> p[g] ;
}
model {
for(i in 1:g){
x[i] ~ binomial(n[i], p[i]) ;
}
}
generated quantities {
vector[g] log_lik ;
vector[g] prob_p_upper_05 ;
for(i in 1:g){
prob_p_upper_05[i] = p[i] > 0.5 ? 1 : 0 ;
log_lik[i] = binomial_lpmf(x[i] | n[i], p[i]) ;
}
}
統計モデルによる事後分布のサンプリング
from IPython.core.display import display import numpy as np import matplotlib import matplotlib.pyplot as plt import os import pandas as pd import pickle import pystan from tabulate import tabulate matplotlib.rcParams['figure.figsize'] = (10, 20) # Stanで使うデータの用意 stan_data = {"g": 1, "x": np.array([55]), "n": np.array([100])} # 興味のあるパラメータの設定 par = ["p", "prob_p_upper_05", "log_lik"] prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975] # モデルの読み込み stan_file_c = os.path.join("stan", "binomial.pkl") with open(stan_file_c, "rb") as f: model = pickle.load(f) # MCMCでサンプリング logger.info("Start MCMC sampling") fit = model.sampling(data=stan_data, pars=par, iter=21000, chains=5, warmup=1000, seed=1234, algorithm="NUTS") # 事後分布の表を取得 summary = fit.summary(pars=par, probs=prob) summary_df = pd.DataFrame(summary["summary"], index=summary["summary_rownames"], columns=summary["summary_colnames"]) display(summary_df) summary_df_path = os.path.join(result_dir, "df_summary_1.md") with open(summary_df_path, "w") as f: f.write(tabulate(summary_df, summary_df.columns, tablefmt="pipe")) logger.info("MCMC result summary is saved at {0}".format(summary_df_path)) # 事後分布の可視化 fit.traceplot(par) traceplot_path = os.path.join(result_dir, "traceplot_1.png") plt.savefig(traceplot_path) plt.show() logger.info("traceplot result is saved at {0}".format(traceplot_path)) # WAICの計算 log_lik = fit.extract("log_lik")["log_lik"] waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0)) logger.info("WAICの値は{0}です".format(waic))
- 結果(表と図)
| mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p[0] | 0.548981 | 0.000259346 | 0.0491617 | 0.451546 | 0.467666 | 0.515895 | 0.549386 | 0.582306 | 0.629074 | 0.644074 | 35933 | 1.00001 |
| prob_p_upper_05[0] | 0.83937 | 0.00175169 | 0.367191 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 43941 | 1.00003 |
| log_lik[0] | -3.02129 | 0.0034005 | 0.708006 | -5.02757 | -4.429 | -3.17868 | -2.74975 | -2.57516 | -2.52779 | -2.52636 | 43350 | 1.00014 |

比率の差の推測
相談相手問題(1つの2項分布)
Stanによる統計モデルの構築
import os import pystan import pickle # Stanのモデルを読み込んでコンパイルする stan_file = os.path.join("stan", "multinomial.stan") stan_file_c = os.path.join("stan", "multinomial.pkl") model = pystan.StanModel(file=stan_file) with open(stan_file_c, "wb") as f: pickle.dump(model, f) logger.info("Stan model is compiled to {0}".format(stan_file_c))
- multinomial.stan
data {
int<lower=0> a;
int<lower=0> b;
int<lower=0> x[a, b] ;
}
transformed data {
# セル数の和
int<lower=0> ab ;
# データのベクトル形式
int<lower=0> xy[a*b] ;
# 値の和
int<lower=0> N ;
ab = a * b ;
for(i in 1:a){
for(j in 1:b){
xy[(i - 1) * b + j] = x[i, j] ;
}
}
# sum関数はint[,]では計算できないので、ベクトル形式に変換が必要
N = sum(xy) ;
}
parameters {
# simplex型は和が1となる
simplex[ab] p ;
}
transformed parameters {
# simplex型はベクトルにしか使えないので、matrix形式に変換する
real<lower=0, upper=1> pm[a, b] ;
for(i in 1:a){
for(j in 1:b){
pm[i, j] = p[(i - 1) * b + j] ;
}
}
}
model {
xy ~ multinomial(p) ;
}
generated quantities{
int<lower=1, upper=ab> index ;
vector[ab] log_lik ;
real pa[a] ;
real pb[b] ;
real pr[ab] ;
real pr2[ab] ;
real cac ;
# 周辺確率の計算
for(i in 1:a){
pa[i] = sum(pm[i, :]) ;
}
for(i in 1:b){
pb[i] = sum(pm[:, i]) ;
}
for(i in 1:a){
for(j in 1:b){
index = (i - 1) * b + j ;
pr[index] = (pm[i, j] - pa[i] * pb[j]) / sqrt(pa[i] * pb[j]) ;
pr2[index] = pow(pr[index], 2) ;
log_lik[index] = multinomial_lpmf(xy | p) ;
}
}
cac = sqrt(sum(pr2) / (min(a, b) - 1)) ;
}
統計モデルによる事後分布のサンプリング
from IPython.core.display import display
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
import os
import pandas as pd
import pickle
import pystan
from tabulate import tabulate
matplotlib.rcParams['figure.figsize'] = (10, 10)
# Stanで使うデータの用意
stan_data = {"a": 1,
"b": 6,
"x": np.array([[30,12,4,20,22,8]])}
# 興味のあるパラメータの設定
par = ["p",
"log_lik"]
prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975]
# モデルの読み込み
stan_file_c = os.path.join("stan", "multinomial.pkl")
with open(stan_file_c, "rb") as f:
model = pickle.load(f)
# MCMCでサンプリング
logger.info("Start MCMC sampling")
fit = model.sampling(data=stan_data,
pars=par,
iter=21000,
chains=5,
warmup=1000,
seed=1234,
algorithm="NUTS")
# 事後分布の表を取得
summary = fit.summary(pars=par, probs=prob)
summary_df = pd.DataFrame(summary["summary"],
index=summary["summary_rownames"],
columns=summary["summary_colnames"])
display(summary_df)
summary_df_path = os.path.join(result_dir, "df_summary_2.md")
with open(summary_df_path, "w") as f:
f.write(tabulate(summary_df, summary_df.columns, tablefmt="pipe"))
logger.info("MCMC result summary is saved at {0}".format(summary_df_path))
# 事後分布の可視化
fit.traceplot(par)
traceplot_path = os.path.join(result_dir, "traceplot_2.png")
plt.savefig(traceplot_path)
plt.show()
logger.info("traceplot result is saved at {0}".format(traceplot_path))
# WAICの計算
log_lik = fit.extract("log_lik")["log_lik"]
waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0))
logger.info("WAICの値は{0}です".format(waic))
- 結果(表と図)
| mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p[0] | 0.303771 | 0.000143133 | 0.0452625 | 0.219397 | 0.231827 | 0.272348 | 0.302484 | 0.333784 | 0.380652 | 0.395746 | 100000 | 0.999974 |
| p[1] | 0.127472 | 0.000103832 | 0.0328345 | 0.0703212 | 0.077966 | 0.104117 | 0.125192 | 0.148024 | 0.185467 | 0.198181 | 100000 | 0.999978 |
| p[2] | 0.0490361 | 6.7299e-05 | 0.0212818 | 0.0162597 | 0.019695 | 0.0335044 | 0.0460529 | 0.0616122 | 0.0882118 | 0.098237 | 100000 | 0.999981 |
| p[3] | 0.206034 | 0.000126221 | 0.0399147 | 0.133482 | 0.143911 | 0.178019 | 0.204084 | 0.231992 | 0.275117 | 0.289446 | 100000 | 0.999987 |
| p[4] | 0.225439 | 0.000129709 | 0.0410175 | 0.150289 | 0.161048 | 0.196605 | 0.223729 | 0.252287 | 0.295423 | 0.310788 | 100000 | 0.999972 |
| p[5] | 0.0882487 | 8.84886e-05 | 0.0279826 | 0.0416277 | 0.0471727 | 0.068063 | 0.0855951 | 0.105674 | 0.138238 | 0.149799 | 100000 | 1.00002 |
| log_lik[0] | -12.4762 | 0.00655559 | 1.51627 | -16.25 | -15.3943 | -13.2568 | -12.1647 | -11.3654 | -10.6288 | -10.4778 | 53497 | 1 |
| log_lik[1] | -12.4762 | 0.00655559 | 1.51627 | -16.25 | -15.3943 | -13.2568 | -12.1647 | -11.3654 | -10.6288 | -10.4778 | 53497 | 1 |
| log_lik[2] | -12.4762 | 0.00655559 | 1.51627 | -16.25 | -15.3943 | -13.2568 | -12.1647 | -11.3654 | -10.6288 | -10.4778 | 53497 | 1 |
| log_lik[3] | -12.4762 | 0.00655559 | 1.51627 | -16.25 | -15.3943 | -13.2568 | -12.1647 | -11.3654 | -10.6288 | -10.4778 | 53497 | 1 |
| log_lik[4] | -12.4762 | 0.00655559 | 1.51627 | -16.25 | -15.3943 | -13.2568 | -12.1647 | -11.3654 | -10.6288 | -10.4778 | 53497 | 1 |
| log_lik[5] | -12.4762 | 0.00655559 | 1.51627 | -16.25 | -15.3943 | -13.2568 | -12.1647 | -11.3654 | -10.6288 | -10.4778 | 53497 | 1 |

治療法問題(2つの2項分布)
Stanによる統計モデルの構築
- binomial.stanをそのまま利用
統計モデルによる事後分布のサンプリング
from IPython.core.display import display import numpy as np import matplotlib import matplotlib.pyplot as plt import os import pandas as pd import pickle import pystan from tabulate import tabulate matplotlib.rcParams['figure.figsize'] = (10, 10) # Stanで使うデータの用意 stan_data = {"g": 2, "x": np.array([90, 78]), "n": np.array([115, 117])} # 興味のあるパラメータの設定 par = ["p", "log_lik"] prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975] # モデルの読み込み stan_file_c = os.path.join("stan", "binomial.pkl") with open(stan_file_c, "rb") as f: model = pickle.load(f) # MCMCでサンプリング logger.info("Start MCMC sampling") fit = model.sampling(data=stan_data, pars=par, iter=21000, chains=5, warmup=1000, seed=1234, algorithm="NUTS") # 事後分布の表を取得 summary = fit.summary(pars=par, probs=prob) summary_df = pd.DataFrame(summary["summary"], index=summary["summary_rownames"], columns=summary["summary_colnames"]) display(summary_df) summary_df_path = os.path.join(result_dir, "df_summary_3.md") with open(summary_df_path, "w") as f: f.write(tabulate(summary_df, summary_df.columns, tablefmt="pipe")) logger.info("MCMC result summary is saved at {0}".format(summary_df_path)) # 事後分布の可視化 fit.traceplot(par) traceplot_path = os.path.join(result_dir, "traceplot_3.png") plt.savefig(traceplot_path) plt.show() logger.info("traceplot result is saved at {0}".format(traceplot_path)) # WAICの計算 log_lik = fit.extract("log_lik")["log_lik"] waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0)) logger.info("WAICの値は{0}です".format(waic))
- 結果(表と図)
| mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p[0] | 0.777804 | 0.000131177 | 0.0381837 | 0.698637 | 0.712412 | 0.752983 | 0.779475 | 0.804413 | 0.837762 | 0.847901 | 84731 | 1.00002 |
| p[1] | 0.663806 | 0.000142741 | 0.0431943 | 0.576869 | 0.591607 | 0.635059 | 0.664743 | 0.693503 | 0.733634 | 0.746176 | 91571 | 1.00004 |
| log_lik[0] | -2.9017 | 0.00319529 | 0.699761 | -4.89146 | -4.30242 | -3.06149 | -2.63201 | -2.45849 | -2.41124 | -2.40982 | 47960 | 1.00004 |
| log_lik[1] | -3.04626 | 0.00319586 | 0.700447 | -5.04176 | -4.45784 | -3.20754 | -2.77576 | -2.60114 | -2.55245 | -2.55097 | 48037 | 0.999998 |

連関の推測
広告効果(2×2のクロス表)
Stanによる統計モデルの構築
- multinomial.stanをそのまま利用
統計モデルによる事後分布のサンプリング
from IPython.core.display import display import numpy as np import matplotlib import matplotlib.pyplot as plt import os import pandas as pd import pickle import pystan from tabulate import tabulate matplotlib.rcParams['figure.figsize'] = (10, 20) # Stanで使うデータの用意 stan_data = {"a": 2, "b": 2, "x": np.array([[33, 18], [84, 23]])} # 興味のあるパラメータの設定 par = ["p", "pr", "cac", "log_lik"] prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975] # モデルの読み込み stan_file_c = os.path.join("stan", "multinomial.pkl") with open(stan_file_c, "rb") as f: model = pickle.load(f) # MCMCでサンプリング logger.info("Start MCMC sampling") fit = model.sampling(data=stan_data, pars=par, iter=21000, chains=5, warmup=1000, seed=1234, algorithm="NUTS") # 事後分布の表を取得 summary = fit.summary(pars=par, probs=prob) summary_df = pd.DataFrame(summary["summary"], index=summary["summary_rownames"], columns=summary["summary_colnames"]) display(summary_df) summary_df_path = os.path.join(result_dir, "df_summary_4.md") with open(summary_df_path, "w") as f: f.write(tabulate(summary_df, summary_df.columns, tablefmt="pipe")) logger.info("MCMC result summary is saved at {0}".format(summary_df_path)) # 事後分布の可視化 fit.traceplot(par) traceplot_path = os.path.join(result_dir, "traceplot_4.png") plt.savefig(traceplot_path) plt.show() logger.info("traceplot result is saved at {0}".format(traceplot_path)) # WAICの計算 log_lik = fit.extract("log_lik")["log_lik"] waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0)) logger.info("WAICの値は{0}です".format(waic))
- 結果(表と図)
| mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p[0] | 0.209943 | 0.000100962 | 0.031927 | 0.151078 | 0.159612 | 0.187721 | 0.208703 | 0.230906 | 0.264185 | 0.275663 | 100000 | 0.999996 |
| p[1] | 0.117181 | 7.95044e-05 | 0.0251415 | 0.072712 | 0.0787064 | 0.0994485 | 0.115658 | 0.133121 | 0.161086 | 0.170502 | 100000 | 0.999968 |
| p[2] | 0.524635 | 0.000123124 | 0.0389353 | 0.448127 | 0.46035 | 0.498185 | 0.524872 | 0.550989 | 0.588352 | 0.600344 | 100000 | 0.999988 |
| p[3] | 0.148241 | 8.82448e-05 | 0.0279055 | 0.0976095 | 0.104724 | 0.128717 | 0.146867 | 0.166217 | 0.196445 | 0.206956 | 100000 | 1.00002 |
| pr[0] | -0.0618101 | 0.000109123 | 0.0345078 | -0.13111 | -0.119605 | -0.0846981 | -0.061201 | -0.0382136 | -0.00607329 | 0.00411544 | 100000 | 0.999975 |
| pr[1] | 0.102741 | 0.000178103 | 0.0563212 | -0.00695254 | 0.0102769 | 0.0642157 | 0.102485 | 0.141141 | 0.195901 | 0.214191 | 100000 | 0.999981 |
| pr[2] | 0.0431673 | 7.75096e-05 | 0.0245107 | -0.0028464 | 0.00422608 | 0.0263984 | 0.0423908 | 0.0590213 | 0.0846067 | 0.0937759 | 100000 | 0.999969 |
| pr[3] | -0.0716558 | 0.000125491 | 0.0396838 | -0.151139 | -0.137544 | -0.0983243 | -0.0712307 | -0.0444078 | -0.00712286 | 0.00476227 | 100000 | 0.999974 |
| cac | 0.148376 | 0.00024284 | 0.0767926 | 0.0130583 | 0.0253949 | 0.0916138 | 0.145924 | 0.200898 | 0.279509 | 0.305541 | 100000 | 0.999981 |
| log_lik[0] | -8.68599 | 0.00539516 | 1.20283 | -11.8121 | -11.0697 | -9.22119 | -8.38001 | -7.80783 | -7.38651 | -7.31759 | 49705 | 1.00009 |
| log_lik[1] | -8.68599 | 0.00539516 | 1.20283 | -11.8121 | -11.0697 | -9.22119 | -8.38001 | -7.80783 | -7.38651 | -7.31759 | 49705 | 1.00009 |
| log_lik[2] | -8.68599 | 0.00539516 | 1.20283 | -11.8121 | -11.0697 | -9.22119 | -8.38001 | -7.80783 | -7.38651 | -7.31759 | 49705 | 1.00009 |
| log_lik[3] | -8.68599 | 0.00539516 | 1.20283 | -11.8121 | -11.0697 | -9.22119 | -8.38001 | -7.80783 | -7.38651 | -7.31759 | 49705 | 1.00009 |

ワイン(3×3のクロス表)
Stanによる統計モデルの構築
- multinomial.stanをそのまま利用
統計モデルによる事後分布のサンプリング
from IPython.core.display import display import numpy as np import matplotlib import matplotlib.pyplot as plt import os import pandas as pd import pickle import pystan from tabulate import tabulate matplotlib.rcParams['figure.figsize'] = (10, 20) # Stanで使うデータの用意 stan_data = {"a": 3, "b": 3, "x": np.array([[13, 6, 21], [7, 17, 7], [13, 6, 13]])} # 興味のあるパラメータの設定 par = ["p", "pr", "cac", "log_lik"] prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975] # モデルの読み込み stan_file_c = os.path.join("stan", "multinomial.pkl") with open(stan_file_c, "rb") as f: model = pickle.load(f) # MCMCでサンプリング logger.info("Start MCMC sampling") fit = model.sampling(data=stan_data, pars=par, iter=21000, chains=5, warmup=1000, seed=1234, algorithm="NUTS") # 事後分布の表を取得 summary = fit.summary(pars=par, probs=prob) summary_df = pd.DataFrame(summary["summary"], index=summary["summary_rownames"], columns=summary["summary_colnames"]) display(summary_df) summary_df_path = os.path.join(result_dir, "df_summary_5.md") with open(summary_df_path, "w") as f: f.write(tabulate(summary_df, summary_df.columns, tablefmt="pipe")) logger.info("MCMC result summary is saved at {0}".format(summary_df_path)) # 事後分布の可視化 fit.traceplot(par) traceplot_path = os.path.join(result_dir, "traceplot_5.png") plt.savefig(traceplot_path) plt.show() logger.info("traceplot result is saved at {0}".format(traceplot_path)) # WAICの計算 log_lik = fit.extract("log_lik")["log_lik"] waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0)) logger.info("WAICの値は{0}です".format(waic))
- 結果(表と図)
| mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p[0] | 0.125075 | 9.88918e-05 | 0.0312723 | 0.0706493 | 0.0778646 | 0.102857 | 0.12275 | 0.14495 | 0.180202 | 0.19217 | 100000 | 0.999969 |
| p[1] | 0.0625879 | 7.2072e-05 | 0.0227912 | 0.0257446 | 0.0299953 | 0.0460044 | 0.0600517 | 0.076303 | 0.104002 | 0.114101 | 100000 | 0.999979 |
| p[2] | 0.1964 | 0.000118333 | 0.0374201 | 0.128527 | 0.137895 | 0.170376 | 0.194506 | 0.220619 | 0.260754 | 0.27471 | 100000 | 0.999964 |
| p[3] | 0.071492 | 7.68005e-05 | 0.0242864 | 0.0314877 | 0.0362726 | 0.053982 | 0.0688749 | 0.0863221 | 0.115217 | 0.12588 | 100000 | 0.99997 |
| p[4] | 0.160706 | 0.000109287 | 0.0345596 | 0.0990021 | 0.107436 | 0.136436 | 0.158562 | 0.182849 | 0.220719 | 0.233882 | 100000 | 0.999966 |
| p[5] | 0.0713786 | 7.66519e-05 | 0.0242395 | 0.0317293 | 0.0362675 | 0.0539307 | 0.0688132 | 0.086041 | 0.115415 | 0.125865 | 100000 | 0.999966 |
| p[6] | 0.124919 | 9.84871e-05 | 0.0311444 | 0.0707794 | 0.0779037 | 0.102729 | 0.122671 | 0.144522 | 0.179687 | 0.191926 | 100000 | 1 |
| p[7] | 0.0625321 | 7.237e-05 | 0.0228854 | 0.025732 | 0.029808 | 0.0459515 | 0.0599421 | 0.0762803 | 0.104219 | 0.114268 | 100000 | 0.999984 |
| p[8] | 0.12491 | 9.85523e-05 | 0.031165 | 0.0709911 | 0.0779175 | 0.102493 | 0.122675 | 0.144806 | 0.179601 | 0.191908 | 100000 | 0.999981 |
| pr[0] | 0.00455472 | 0.000191147 | 0.0604459 | -0.111419 | -0.0932988 | -0.0370003 | 0.00381112 | 0.0454738 | 0.104957 | 0.123828 | 100000 | 0.99997 |
| pr[1] | -0.141598 | 0.000173091 | 0.0547361 | -0.240132 | -0.226578 | -0.180169 | -0.144331 | -0.106119 | -0.046784 | -0.026671 | 100000 | 0.999976 |
| pr[2] | 0.117052 | 0.000180329 | 0.0570251 | 0.00421428 | 0.0226175 | 0.0784596 | 0.117427 | 0.155714 | 0.210575 | 0.22766 | 100000 | 0.999961 |
| pr[3] | -0.0830821 | 0.00018874 | 0.059685 | -0.19199 | -0.176676 | -0.125273 | -0.0857259 | -0.0437753 | 0.019607 | 0.0406338 | 100000 | 0.99999 |
| pr[4] | 0.250214 | 0.000209959 | 0.066395 | 0.116994 | 0.139032 | 0.20578 | 0.251421 | 0.295633 | 0.357572 | 0.377051 | 100000 | 0.999979 |
| pr[5] | -0.137782 | 0.000173256 | 0.0547885 | -0.237001 | -0.223096 | -0.176572 | -0.140576 | -0.102 | -0.0427316 | -0.0240325 | 100000 | 0.999979 |
| pr[6] | 0.077024 | 0.000205633 | 0.065027 | -0.048242 | -0.0291772 | 0.0322187 | 0.0765335 | 0.121479 | 0.184749 | 0.204917 | 100000 | 1.00001 |
| pr[7] | -0.0890021 | 0.000190123 | 0.0601222 | -0.197285 | -0.182537 | -0.131403 | -0.0920298 | -0.0497219 | 0.0150218 | 0.0371228 | 100000 | 0.999977 |
| pr[8] | 0.00640808 | 0.000190191 | 0.0601436 | -0.108758 | -0.0916682 | -0.0350148 | 0.0061111 | 0.0472022 | 0.106246 | 0.125763 | 100000 | 0.999969 |
| cac | 0.283669 | 0.000194402 | 0.0614752 | 0.162228 | 0.181986 | 0.242123 | 0.28426 | 0.325619 | 0.384129 | 0.402499 | 100000 | 0.999977 |
| log_lik[0] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[1] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[2] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[3] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[4] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[5] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[6] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[7] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |
| log_lik[8] | -19.3975 | 0.00872255 | 1.87604 | -23.8484 | -22.9175 | -20.4394 | -19.0998 | -18.0235 | -16.9287 | -16.6699 | 46259 | 1.00004 |

備考
個人的には、クロス集計はよく使いますのでここで扱ってくれたのはいいものだと思いました。
- ただクロス集計では次のような問題には対応できない
- A群とB群の人間が居て、それぞれa人とb人存在する
- 各人は好きな色(全部でn種類)の飴を好きなだけピックアップする
- 各人の飴の本数、m本は任意の値をとる
- この時、A群とB群で特定の色の飴が取得されやすいか、されづらいかはどう判定すればよい?
- こういう問題ですと、より階層的なモデルが必要になってきます。ぱっと思いつくのはディリクレ分布を使って、トピックの混合率をモデル化するトピックモデルを使う手法でしょうか。
- ただクロス集計では次のような問題には対応できない
「はじめての統計データ分析」はこれで最終回ですね。大分Stanへの恐怖心もなくなってきましたので、次は「実践ベイズモデリング」とか「StanとRでベイズ統計モデリング」あたりを流していこうかと思います。
はじめての 統計データ分析 ―ベイズ的〈ポストp値時代〉の統計学― その6
その6です。今回は第5章の章末問題に取り組んでいきます。
5章 実験計画による多群の差の推測
内容
- 分散分析によるF検定へのオルタナ
- 要因から生じる影響の調査
- 要因 : 離散的なカテゴリーによる影響のこと
- 水準: 要因が取りうる様々な状態のこと
- 水準数: 水準のパターン数。以下要因
の水準数を
で表す。
独立した1要因モデル
- モデル
- 要因の
番目の水準における
番目の測定値が、
番目の水準がもつ平均的な効果と正規分布から生じる乱数による現れる
- 以上を纏めて、事後確率は
と表される
- 水準内の測定値は独立に観測されてると考える
- 同時確率は
- 即ち、尤度は
- 同時確率は
- モデル
生成量
モデルを使った要因の効果の方法例
独立した2要因モデル
- 要因を複数導入
- 水準の組み合わせ1単位のことをセルと以下では呼称する
- 交互作用: 一方の要因の水準毎に、他方の要因の平均の高低パターンが異なる現象
- モデル
-
-
- 水準
の
番目の測定値が、全体の平均に要因
の
番目の水準の効果 + 要因
の
番目の水準の効果 +
と
の交互作用の効果 + 乱数から構成されることを示す
- セルの平均 + 乱数
-
- それぞれ全平均(水準の効果)の性質から次の制約を持つ(水準の効果を全水準で足すと0になる性質を定式化)
- 尤度
- 要因を複数導入
章末問題
1要因の推測
データの取得
import numpy as np import os from logging import getLogger, Formatter, StreamHandler, DEBUG # printではなくloggerを使う def get_logger(): logger = getLogger(__name__) logger.setLevel(DEBUG) log_fmt = '%(asctime)s : %(name)s : %(levelname)s : %(message)s' formatter = Formatter(log_fmt) stream_handler = StreamHandler() stream_handler.setLevel(DEBUG) stream_handler.setFormatter(formatter) logger.addHandler(stream_handler) return logger logger = get_logger() # データの準備 ld = np.array([5.02, 6.67, 8.17, 2.79, 8.13, 6.34, 6.32, 3.97]) ll = np.array([9.89, 9.58, 11.20, 9.05, 12.33, 9.39, 10.88, 9.37, 17.40]) dm = np.array([10.20, 7.29, 7.57, 3.42, 5.82, 10.92, 5.21, 13.47, 8.64, 6.05]) y = np.concatenate([a, b, c]) A_ld = np.full(ld.size, 1, np.int) A_ll = np.full(ll.size, 2, np.int) A_dm = np.full(dm.size, 3, np.int) A = np.concatenate([A_ld, A_ll, A_dm]) # 出力用ディレクトリの用意 result_dir = os.path.join("result", "chapter5") if os.path.exists(result_dir) is False: os.makedirs(result_dir) logger.info("{0} is made".format(result_dir))
Stanによる統計モデルの構築
特に難しいところは無いような気がします。説明量を計算するために、分散値を計算する手間があるぐらいでしょうか?
import os import pystan import pickle # Stanのモデルを読み込んでコンパイルする stan_file = os.path.join("stan", "e1_ind.stan") stan_file_c = os.path.join("stan", "e1_ind.pkl") model = pystan.StanModel(file=stan_file) with open(stan_file_c, "wb") as f: pickle.dump(model, f) logger.info("Stan model is compiled to {0}".format(stan_file_c))
e1_ind.stan
data {
int<lower=0> n ;
int<lower=0> a ;
real<lower=0> y[n] ;
int<lower=1, upper=3> A[n] ;
}
parameters {
vector[a] mu_a ;
real<lower=0> sigma_e ;
}
model {
for(i in 1:n){
y[i] ~ normal(mu_a[A[i]], sigma_e) ;
}
}
generated quantities {
real mu ;
vector[a] eol ;
vector[a] eol2 ;
vector[a] prob_eol_upper_0 ;
real var_e ;
real var_a ;
real eta2 ;
real delta ;
int<lower=0, upper=1> prob_ll_upper_dm ;
int<lower=0, upper=1> prob_ll_upper_ld ;
int<lower=0, upper=1> prob_dm_upper_ld ;
vector[n] log_lik ;
mu = mean(mu_a) ;
for(i in 1:a){
eol[i] = mu_a[i] - mu ;
eol2[i] = pow(eol[i], 2) ;
prob_eol_upper_0[i] = eol[i] > 0 ? 1 : 0 ;
}
var_a = mean(eol2) ;
var_e = pow(sigma_e, 2) ;
eta2 = var_a / (var_a + var_e) ;
delta = sqrt(var_a) / sigma_e ;
prob_ll_upper_dm = mu_a[2] - mu_a[3] > 0 ? 1 : 0 ;
prob_ll_upper_ld = mu_a[2] - mu_a[1] > 0 ? 1 : 0 ;
prob_dm_upper_ld = mu_a[3] - mu_a[1] > 0 ? 1 : 0 ;
for(i in 1:n){
log_lik[i] = normal_lpdf(y[i] | mu_a[A[i]], sigma_e) ;
}
}
統計モデルによる事後分布のサンプリング
import pandas as pd import pickle import pystan import matplotlib import matplotlib.pyplot as plt import os from IPython.core.display import display from tabulate import tabulate matplotlib.rcParams['figure.figsize'] = (10, 50) # Stanで使うデータの用意 stan_data = {"n": y.size, "a": 3, "y": y, "A": A} # 興味のあるパラメータの設定 par = ["mu_a", "sigma_e", "eol", "eta2", "delta", "prob_eol_upper_0", "prob_ll_upper_dm", "prob_ll_upper_ld", "prob_dm_upper_ld", "log_lik"] prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975] # モデルの読み込み stan_file_c = os.path.join("stan", "e1_ind.pkl") with open(stan_file_c, "rb") as f: model = pickle.load(f) # MCMCでサンプリング logger.info("Start MCMC sampling") fit = model.sampling(data=stan_data, pars=par, iter=21000, chains=5, warmup=1000, seed=1234, algorithm="NUTS") # 事後分布の表を取得 summary = fit.summary(pars=par, probs=prob) summary_df = pd.DataFrame(summary["summary"], index=summary["summary_rownames"], columns=summary["summary_colnames"]) display(summary_df) summary_df_path = os.path.join(result_dir, "df_summary_1.md") with open(summary_df_path, "w") as f: f.write(tabulate(summary_df, summary_df.columns, tablefmt="pipe")) logger.info("MCMC result summary is saved at {0}".format(summary_df_path)) # 事後分布の可視化 fit.traceplot(par) traceplot_path = os.path.join(result_dir, "traceplot_1.png") plt.savefig(traceplot_path) plt.show() logger.info("traceplot result is saved at {0}".format(traceplot_path)) # WAICの計算 log_lik = fit.extract("log_lik")["log_lik"] waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0)) logger.info("WAICの値は{0}です".format(waic))
- 結果(表と分布)
| mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mu_a[0] | 5.92465 | 0.0030795 | 0.973823 | 3.99253 | 4.32772 | 5.28835 | 5.92998 | 6.56208 | 7.51919 | 7.84787 | 100000 | 1.00007 |
| mu_a[1] | 11.0072 | 0.00292346 | 0.924478 | 9.17396 | 9.49254 | 10.4026 | 11.0076 | 11.6092 | 12.5191 | 12.8357 | 100000 | 0.999985 |
| mu_a[2] | 7.85955 | 0.00276635 | 0.874797 | 6.12594 | 6.42644 | 7.28474 | 7.86113 | 8.43353 | 9.29189 | 9.58305 | 100000 | 1 |
| sigma_e | 2.74451 | 0.00151073 | 0.42629 | 2.06146 | 2.14526 | 2.44184 | 2.69385 | 2.9907 | 3.51953 | 3.72462 | 79623 | 0.999968 |
| eol[0] | -2.33916 | 0.00245045 | 0.7749 | -3.88038 | -3.61524 | -2.8447 | -2.33923 | -1.83011 | -1.07629 | -0.817259 | 100000 | 1.00008 |
| eol[1] | 2.74342 | 0.00239336 | 0.756846 | 1.24128 | 1.50518 | 2.24837 | 2.74077 | 3.23758 | 3.98729 | 4.24112 | 100000 | 0.999999 |
| eol[2] | -0.404261 | 0.00233393 | 0.738053 | -1.86101 | -1.60891 | -0.887243 | -0.405952 | 0.0805197 | 0.804455 | 1.05156 | 100000 | 1.00003 |
| eta2 | 0.384362 | 0.000399327 | 0.126278 | 0.122444 | 0.162176 | 0.299146 | 0.391642 | 0.476528 | 0.579842 | 0.608479 | 100000 | 0.999981 |
| delta | 0.804616 | 0.000704594 | 0.222812 | 0.373535 | 0.439964 | 0.653323 | 0.802353 | 0.954108 | 1.17476 | 1.24665 | 100000 | 0.999984 |
| prob_eol_upper_0[0] | 0.00215 | 0.000165199 | 0.0463185 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 78613 | 1.00006 |
| prob_eol_upper_0[1] | 0.99963 | 6.2415e-05 | 0.0192319 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 94944 | 1.00003 |
| prob_eol_upper_0[2] | 0.28713 | 0.00152318 | 0.452425 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 88225 | 1.00002 |
| prob_ll_upper_dm | 0.99168 | 0.000333967 | 0.0908342 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 73976 | 0.999978 |
| prob_ll_upper_ld | 0.99975 | 5.19414e-05 | 0.0158095 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 92642 | 0.999991 |
| prob_dm_upper_ld | 0.93277 | 0.000930336 | 0.250421 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 72454 | 1.00007 |
| log_lik[0] | -2.03678 | 0.000758605 | 0.199372 | -2.49373 | -2.39575 | -2.14793 | -2.01548 | -1.89931 | -1.75258 | -1.7086 | 69071 | 0.999994 |
| log_lik[1] | -2.01783 | 0.000754794 | 0.191828 | -2.4532 | -2.35815 | -2.12696 | -1.99886 | -1.88578 | -1.74113 | -1.69764 | 64590 | 1.00002 |
| log_lik[2] | -2.33612 | 0.000992587 | 0.313883 | -3.08375 | -2.92534 | -2.50565 | -2.2841 | -2.11391 | -1.92039 | -1.86752 | 100000 | 1.00004 |
| log_lik[3] | -2.67679 | 0.00135735 | 0.429231 | -3.69657 | -3.47937 | -2.91461 | -2.60959 | -2.36613 | -2.10742 | -2.04192 | 100000 | 1.00004 |
| log_lik[4] | -2.32348 | 0.000978475 | 0.309421 | -3.06092 | -2.90399 | -2.49026 | -2.27211 | -2.10467 | -1.91389 | -1.86127 | 100000 | 1.00004 |
| log_lik[5] | -1.99069 | 0.000735103 | 0.179753 | -2.39033 | -2.3073 | -2.0972 | -1.97534 | -1.86648 | -1.72545 | -1.6824 | 59794 | 1.00001 |
| log_lik[6] | -1.98954 | 0.00073424 | 0.17923 | -2.38764 | -2.30472 | -2.09579 | -1.9742 | -1.86567 | -1.72457 | -1.6816 | 59586 | 1.00001 |
| log_lik[7] | -2.25015 | 0.000893585 | 0.282577 | -2.92974 | -2.78158 | -2.40036 | -2.20673 | -2.05154 | -1.87364 | -1.82393 | 100000 | 1.00003 |
| log_lik[8] | -2.06094 | 0.000756641 | 0.203617 | -2.52871 | -2.42581 | -2.17552 | -2.0385 | -1.91954 | -1.77099 | -1.72765 | 72418 | 1 |
| log_lik[9] | -2.11696 | 0.000790546 | 0.223723 | -2.63793 | -2.52526 | -2.2399 | -2.08855 | -1.96137 | -1.80487 | -1.76051 | 80088 | 0.999999 |
| log_lik[10] | -1.9749 | 0.000705525 | 0.171411 | -2.3497 | -2.27598 | -2.07886 | -1.96221 | -1.85611 | -1.71601 | -1.67376 | 59027 | 1.00001 |
| log_lik[11] | -2.24435 | 0.000846443 | 0.267669 | -2.87634 | -2.74126 | -2.38938 | -2.20473 | -2.05568 | -1.88209 | -1.83201 | 100000 | 0.999994 |
| log_lik[12] | -2.09647 | 0.000768147 | 0.216744 | -2.60167 | -2.49056 | -2.21654 | -2.06987 | -1.94642 | -1.79251 | -1.74675 | 79617 | 1 |
| log_lik[13] | -2.15804 | 0.000816763 | 0.238127 | -2.71684 | -2.59706 | -2.2877 | -2.12586 | -1.9917 | -1.83017 | -1.78421 | 85001 | 0.999997 |
| log_lik[14] | -1.97342 | 0.000706486 | 0.170781 | -2.34448 | -2.27222 | -2.07722 | -1.96171 | -1.85505 | -1.71519 | -1.67338 | 58435 | 1.00001 |
| log_lik[15] | -2.16266 | 0.000819785 | 0.239732 | -2.72544 | -2.60451 | -2.29303 | -2.13016 | -1.99503 | -1.83272 | -1.78664 | 85517 | 0.999997 |
| log_lik[16] | -4.87403 | 0.00339268 | 1.07286 | -7.32982 | -6.8243 | -5.5138 | -4.74535 | -4.09518 | -3.35255 | -3.15931 | 100000 | 0.999977 |
| log_lik[17] | -2.35558 | 0.000919091 | 0.290642 | -3.03811 | -2.89536 | -2.51793 | -2.31435 | -2.14877 | -1.95975 | -1.90793 | 100000 | 1 |
| log_lik[18] | -1.98969 | 0.000680232 | 0.173859 | -2.37117 | -2.2949 | -2.09513 | -1.97689 | -1.86882 | -1.72784 | -1.68611 | 65325 | 0.999965 |
| log_lik[19] | -1.97261 | 0.000674084 | 0.168196 | -2.33564 | -2.26748 | -2.07642 | -1.96134 | -1.85564 | -1.7164 | -1.67546 | 62259 | 0.999965 |
| log_lik[20] | -3.36627 | 0.00188208 | 0.595165 | -4.73815 | -4.46265 | -3.7144 | -3.2838 | -2.93321 | -2.54231 | -2.44062 | 100000 | 0.999985 |
| log_lik[21] | -2.26206 | 0.000825123 | 0.260927 | -2.87464 | -2.74387 | -2.40867 | -2.22729 | -2.07788 | -1.90249 | -1.85212 | 100000 | 0.999979 |
| log_lik[22] | -2.6317 | 0.00118696 | 0.37535 | -3.50938 | -3.32991 | -2.84462 | -2.57612 | -2.36118 | -2.12327 | -2.05933 | 100000 | 1 |
| log_lik[23] | -2.46518 | 0.00102346 | 0.323646 | -3.2244 | -3.0644 | -2.64582 | -2.41881 | -2.23303 | -2.02502 | -1.96827 | 100000 | 0.999983 |
| log_lik[24] | -4.20173 | 0.00267471 | 0.845818 | -6.14049 | -5.74926 | -4.70266 | -4.09605 | -3.58666 | -3.0153 | -2.86741 | 100000 | 0.99999 |
| log_lik[25] | -2.00989 | 0.000689887 | 0.180727 | -2.40828 | -2.32687 | -2.11763 | -1.99477 | -1.88481 | -1.74166 | -1.69933 | 68626 | 0.999981 |
| log_lik[26] | -2.19919 | 0.000762851 | 0.241235 | -2.76133 | -2.64207 | -2.33492 | -2.16884 | -2.03002 | -1.86336 | -1.81479 | 100000 | 0.999977 |

2要因の推測
データの取得
データの取得がめちゃめちゃ汚い気がしますが、許してください…
import numpy as np import os from logging import getLogger, Formatter, StreamHandler, DEBUG # printではなくloggerを使う def get_logger(): logger = getLogger(__name__) logger.setLevel(DEBUG) log_fmt = '%(asctime)s : %(name)s : %(levelname)s : %(message)s' formatter = Formatter(log_fmt) stream_handler = StreamHandler() stream_handler.setLevel(DEBUG) stream_handler.setFormatter(formatter) logger.addHandler(stream_handler) return logger logger = get_logger() # データの準備 y = np.array([ 140,146,149,136,147,147,143,143,143,141, 139,136,136,140,135,132,140,134, 123,127,131,130,138,128,129, 115,120,118,118,121,124,129,119,128, 128,124,123,121,122,126,131,122, 121,121,120,116,117,113,118, 143,141,142,145,149,145,143,141,142,155, 138,134,142,136,135,136,131,133, 131,128,128,128,127,130,130, 117,125,132,122,119,122,129,117,127, 117,120,124,122,122,122,118,122, 119,125,122,116,119,113,122]) A_loaded = np.full(49, 1, np.int) A_empty = np.full(y.size - A_loaded.size, 2, np.int) A = np.concatenate([A_loaded, A_empty]) B_loaded_st = np.full(10, 1, np.int) B_loaded_cut = np.full(8, 2, np.int) B_loaded_f = np.full(7, 3, np.int) B_loaded_ch = np.full(9, 4, np.int) B_loaded_sl = np.full(8, 5, np.int) B_loaded_cur = np.full(7, 6, np.int) B_empty_st = np.full(10, 1, np.int) B_empty_cut = np.full(8, 2, np.int) B_empty_f = np.full(7, 3, np.int) B_empty_ch = np.full(9, 4, np.int) B_empty_sl = np.full(8, 5, np.int) B_empty_cur = np.full(7, 6, np.int) B = np.concatenate([B_loaded_st, B_loaded_cut, B_loaded_f, B_loaded_ch, B_loaded_sl, B_loaded_cur, B_empty_st, B_empty_cut, B_empty_f, B_empty_ch, B_empty_sl, B_empty_cur]) # 出力用ディレクトリの用意 result_dir = os.path.join("result", "chapter5") if os.path.exists(result_dir) is False: os.makedirs(result_dir) logger.info("{0} is made".format(result_dir))
Stanによる統計モデルの構築
import os import pystan import pickle # Stanのモデルを読み込んでコンパイルする stan_file = os.path.join("stan", "e2_ind.stan") stan_file_c = os.path.join("stan", "e2_ind.pkl") model = pystan.StanModel(file=stan_file) with open(stan_file_c, "wb") as f: pickle.dump(model, f) logger.info("Stan model is compiled to {0}".format(stan_file_c))
e2_ind.stan
- PyStanがmatrixを可視化する際にうまくうけとってくれないので、ベクトルに変換してからPythonへ渡しています
- 本の付録のStanコードではtransformed parametersの部分が冗長だったので、forとif文で対応しています。
data {
int<lower=1> n ;
int<lower=1> n_a ;
int<lower=1> n_b ;
int<lower=0> y[n] ;
int<lower=1, upper=n_a> A[n] ;
int<lower=1, upper=n_b> B[n] ;
}
parameters {
real<lower=0> mu ;
vector[n_a-1] a ;
vector[n_b-1] b ;
matrix[n_a-1, n_b-1] ab ;
real<lower=0> sigma_e ;
}
transformed parameters {
# 水準の効果の和が0になる制約条件を使う
vector[n_a] a_r ;
vector[n_b] b_r ;
matrix[n_a, n_b] ab_r ;
vector[n_a*n_b] ab_v ;
for(i in 1:n_a-1){
a_r[i] = a[i] ;
}
a_r[n_a] = - sum(a_r[1:n_a - 1]) ;
for(i in 1:(n_b-1)){
b_r[i] = b[i] ;
}
b_r[n_b] = - sum(b_r[1:n_b - 1]) ;
for (j in 1:n_a) {
for (k in 1:n_b) {
if (j == n_a && k == n_b) {
ab_r[j, k] = sum(ab[1:j-1, 1:k-1]) ;
} else if (j == n_a){
ab_r[j, k] = -sum(ab[1:j-1, k]) ;
} else if (k == n_b){
ab_r[j, k] = -sum(ab[j, 1:k-1]) ;
} else {
ab_r[j, k] = ab[j, k] ;
}
ab_v[(j-1) * n_b + k] = ab_r[j, k] ;
}
}
}
model {
for (i in 1:n) {
y[i] ~ normal(mu + a_r[A[i]] + b_r[B[i]] + ab_r[A[i], B[i]], sigma_e) ;
}
}
generated quantities {
vector[n] log_lik ;
vector[n_b] prob_b_upper_0 ;
vector[n_a] a_r2 ;
vector[n_b] b_r2 ;
vector[n_a*n_b] ab_r2 ;
real var_a ;
real var_b ;
real var_ab ;
real var_e ;
real var_y ;
real eta_a2 ;
real eta_b2 ;
real eta_ab2 ;
real eta_t2 ;
real delta_a ;
real delta_b ;
real delta_ab ;
matrix[n_b, n_b] prob_b_comparison ;
for (i in 1:n) {
log_lik[i] = normal_lpdf(y[i] | mu + a_r[A[i]] + b_r[B[i]] + ab_r[A[i], B[i]], sigma_e) ;
}
for (i in 1:n_b) {
prob_b_upper_0[i] = b_r[i] > 0 ? 1 : 0 ;
}
for (i in 1:n_a) {
a_r2[i] = pow(a_r[i], 2) ;
}
for (i in 1:n_b) {
b_r2[i] = pow(b_r[i], 2) ;
}
for (i in 1:n_a*n_b) {
ab_r2[i] = pow(ab_v[i], 2) ;
}
var_a = mean(a_r2) ;
var_b = mean(b_r2) ;
var_ab = mean(ab_r2) ;
var_e = pow(sigma_e, 2) ;
var_y = var_a + var_b + var_ab + var_e ;
eta_a2 = var_a / var_y ;
eta_b2 = var_b / var_y ;
eta_ab2 = var_ab / var_y ;
eta_t2 = (var_a + var_b + var_ab) / var_y ;
delta_a = sqrt(var_a) / sigma_e ;
delta_b = sqrt(var_b) / sigma_e ;
delta_ab = sqrt(var_ab) / sigma_e ;
for (j in 1:n_b){
for (k in 1:n_b){
prob_b_comparison[j, k] = b_r[j] > b_r[k] ? 1 : 0 ;
}
}
}
統計モデルによる事後分布のサンプリング
- データ型が行列のパラメータは、可視化する前に除いています
- 水準の効果の差については、別途ピボットテーブルを作成して可視化しました。
import pandas as pd import pickle import pystan import matplotlib import matplotlib.pyplot as plt import os from IPython.core.display import display from tabulate import tabulate matplotlib.rcParams['figure.figsize'] = (10, 50) # Stanで使うデータの用意 stan_data = {"n": y.size, "n_a": np.unique(A).size, "n_b": np.unique(B).size, "y": y, "A": A, "B": B} # 興味のあるパラメータの設定 pars = ["mu", "sigma_e", "a_r", "b_r", "ab_v", "prob_b_upper_0", "eta_a2", "eta_b2", "eta_ab2", "eta_t2", "delta_a", "delta_b", "delta_ab", "prob_b_comparison", "log_lik"] prob = [0.025, 0.05, 0.25, 0.5, 0.75, 0.95, 0.975] # モデルの読み込み stan_file_c = os.path.join("stan", "e2_ind.pkl") with open(stan_file_c, "rb") as f: model = pickle.load(f) # MCMCでサンプリング logger.info("Start MCMC sampling") fit = model.sampling(data=stan_data, pars=pars, iter=21000, chains=5, warmup=1000, seed=1234, algorithm="NUTS") # 事後分布の表を取得 summary = fit.summary(pars=pars, probs=prob) summary_df = pd.DataFrame(summary["summary"], index=summary["summary_rownames"], columns=summary["summary_colnames"]) display(summary_df) summary_df_path = os.path.join(result_dir, "df_summary_2.md") with open(summary_df_path, "w") as f: f.write(tabulate(summary_df, summary_df.columns, tablefmt="pipe")) logger.info("MCMC result summary is saved at {0}".format(summary_df_path)) # 水準毎に上回る確率を調べる comparison = fit.summary(pars=["prob_b_comparison"], probs=prob) comparison_df = pd.DataFrame(comparison["summary"], index=comparison["summary_rownames"], columns=comparison["summary_colnames"]) comparison_index = pd.Series(comparison_df.index.str.strip("prob_b_comparison[").str.strip("]").str.split(",")) b_dict = {"0":"straight", "1":"cutball", "2":"fork", "3":"changeup", "4":"slider", "5":"curve"} upper_index = comparison_index.apply(lambda x:b_dict[x[0]]) lower_index = comparison_index.apply(lambda x:b_dict[x[1]]) comparison_triple = pd.DataFrame([upper_index, lower_index, comparison_df["mean"].values]).T comparison_triple.columns = ["upper", "lower", "possibility"] comparison_pivot = comparison_triple.pivot(index="upper", columns="lower", values="possibility") display(comparison_pivot) b_df_path = os.path.join(result_dir, "b_comparison.md") with open(b_df_path, "w") as f: f.write(tabulate(comparison_pivot, comparison_pivot.columns, tablefmt="pipe")) logger.info("comparison table is saved at {0}".format(b_df_path)) # 事後分布の可視化 pars.remove("prob_b_upper_0") pars.remove("prob_b_comparison") fit.traceplot(pars) traceplot_path = os.path.join(result_dir, "traceplot_2.png") plt.savefig(traceplot_path) plt.show() logger.info("traceplot result is saved at {0}".format(traceplot_path)) # WAICの計算 log_lik = fit.extract("log_lik")["log_lik"] waic = -2 * np.sum(np.log(np.mean(np.exp(log_lik), axis=0))) + 2 * np.sum(np.var(log_lik, axis=0)) logger.info("WAICの値は{0}です".format(waic))
- 結果(表と分布)
| mean | se_mean | sd | 2.5% | 5% | 25% | 50% | 75% | 95% | 97.5% | n_eff | Rhat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| mu | 128.842 | 0.00125739 | 0.397622 | 128.059 | 128.188 | 128.577 | 128.842 | 129.107 | 129.495 | 129.622 | 100000 | 0.999982 |
| sigma_e | 3.89006 | 0.000955725 | 0.302227 | 3.35328 | 3.42584 | 3.67831 | 3.87124 | 4.08093 | 4.41686 | 4.53412 | 100000 | 1.00008 |
| a_r[0] | 0.0566725 | 0.00125659 | 0.39737 | -0.722588 | -0.595841 | -0.208101 | 0.0562403 | 0.321384 | 0.710725 | 0.840036 | 100000 | 0.999989 |
| a_r[1] | -0.0566725 | 0.00125659 | 0.39737 | -0.840036 | -0.710725 | -0.321384 | -0.0562403 | 0.208101 | 0.595841 | 0.722588 | 100000 | 0.999989 |
| b_r[0] | 15.2089 | 0.00256537 | 0.811242 | 13.6165 | 13.8763 | 14.6635 | 15.2074 | 15.7524 | 16.5397 | 16.8083 | 100000 | 0.999994 |
| b_r[1] | 7.22147 | 0.00280979 | 0.888533 | 5.47369 | 5.76311 | 6.62748 | 7.22389 | 7.81345 | 8.68103 | 8.97568 | 100000 | 1 |
| b_r[2] | 0.303162 | 0.00295644 | 0.93491 | -1.53609 | -1.2345 | -0.321006 | 0.298546 | 0.928776 | 1.83405 | 2.15155 | 100000 | 0.999977 |
| b_r[3] | -6.50771 | 0.00270552 | 0.85556 | -8.17839 | -7.90278 | -7.08083 | -6.51271 | -5.93491 | -5.10512 | -4.82134 | 100000 | 0.999979 |
| b_r[4] | -6.09233 | 0.00281005 | 0.888616 | -7.83205 | -7.54945 | -6.68611 | -6.09106 | -5.49716 | -4.62802 | -4.3491 | 100000 | 0.999976 |
| b_r[5] | -10.1335 | 0.00296533 | 0.93772 | -11.9753 | -11.6719 | -10.7628 | -10.1342 | -9.49813 | -8.59766 | -8.30235 | 100000 | 0.999964 |
| ab_v[0] | -0.607631 | 0.00258641 | 0.817893 | -2.21712 | -1.9559 | -1.15031 | -0.607124 | -0.0654354 | 0.744995 | 1.00949 | 100000 | 0.999985 |
| ab_v[1] | 0.381475 | 0.00281664 | 0.8907 | -1.37495 | -1.08112 | -0.212969 | 0.382057 | 0.976252 | 1.84594 | 2.13356 | 100000 | 0.999998 |
| ab_v[2] | 0.23221 | 0.00297549 | 0.940934 | -1.60767 | -1.31047 | -0.399151 | 0.229995 | 0.866498 | 1.77931 | 2.08061 | 100000 | 0.999969 |
| ab_v[3] | -1.05319 | 0.00268952 | 0.8505 | -2.72909 | -2.45038 | -1.62572 | -1.05216 | -0.482988 | 0.345127 | 0.618895 | 100000 | 0.99998 |
| ab_v[4] | 1.81474 | 0.00281049 | 0.888756 | 0.0653521 | 0.350221 | 1.22523 | 1.81277 | 2.40855 | 3.27038 | 3.56064 | 100000 | 0.999986 |
| ab_v[5] | -0.767603 | 0.00297959 | 0.94223 | -2.60335 | -2.31877 | -1.40147 | -0.769324 | -0.130914 | 0.778092 | 1.07769 | 100000 | 0.999986 |
| ab_v[6] | 0.607631 | 0.00258641 | 0.817893 | -1.00949 | -0.744995 | 0.0654354 | 0.607124 | 1.15031 | 1.9559 | 2.21712 | 100000 | 0.999985 |
| ab_v[7] | -0.381475 | 0.00281664 | 0.8907 | -2.13356 | -1.84594 | -0.976252 | -0.382057 | 0.212969 | 1.08112 | 1.37495 | 100000 | 0.999998 |
| ab_v[8] | -0.23221 | 0.00297549 | 0.940934 | -2.08061 | -1.77931 | -0.866498 | -0.229995 | 0.399151 | 1.31047 | 1.60767 | 100000 | 0.999969 |
| ab_v[9] | 1.05319 | 0.00268952 | 0.8505 | -0.618895 | -0.345127 | 0.482988 | 1.05216 | 1.62572 | 2.45038 | 2.72909 | 100000 | 0.99998 |
| ab_v[10] | -1.81474 | 0.00281049 | 0.888756 | -3.56064 | -3.27038 | -2.40855 | -1.81277 | -1.22523 | -0.350221 | -0.0653521 | 100000 | 0.999986 |
| ab_v[11] | 0.767603 | 0.00297959 | 0.94223 | -1.07769 | -0.778092 | 0.130914 | 0.769324 | 1.40147 | 2.31877 | 2.60335 | 100000 | 0.999986 |
| prob_b_upper_0[0] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_upper_0[1] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_upper_0[2] | 0.62685 | 0.00152942 | 0.483644 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 100000 | 1 |
| prob_b_upper_0[3] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_upper_0[4] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_upper_0[5] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| eta_a2 | 0.00168409 | 1.10406e-05 | 0.00242859 | 1.69709e-06 | 6.70442e-06 | 0.000168269 | 0.000753203 | 0.00220135 | 0.00650296 | 0.00855088 | 48386 | 1.00007 |
| eta_b2 | 0.820698 | 8.31697e-05 | 0.0263006 | 0.762582 | 0.77391 | 0.804681 | 0.822937 | 0.83927 | 0.859708 | 0.865594 | 100000 | 1.00006 |
| eta_ab2 | 0.0179692 | 3.28066e-05 | 0.00948945 | 0.00401563 | 0.00533333 | 0.0109958 | 0.0164943 | 0.0233222 | 0.0357468 | 0.0404685 | 83668 | 1.00012 |
| eta_t2 | 0.840352 | 7.44251e-05 | 0.0235353 | 0.788162 | 0.798497 | 0.826116 | 0.842443 | 0.857014 | 0.875117 | 0.880387 | 100000 | 1.00003 |
| delta_a | 0.0820056 | 0.000277709 | 0.0621218 | 0.0032616 | 0.00655807 | 0.032731 | 0.0691728 | 0.117879 | 0.201804 | 0.231203 | 50039 | 1.00011 |
| delta_b | 2.28756 | 0.000631375 | 0.199658 | 1.90128 | 1.96222 | 2.15243 | 2.28528 | 2.42122 | 2.62019 | 2.68599 | 100000 | 1.00004 |
| delta_ab | 0.325882 | 0.000285382 | 0.0902456 | 0.157327 | 0.181951 | 0.262588 | 0.3238 | 0.386283 | 0.478362 | 0.50898 | 100000 | 1.00008 |
| prob_b_comparison[0,0] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[1,0] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[2,0] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[3,0] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[4,0] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[5,0] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[0,1] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[1,1] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[2,1] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[3,1] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[4,1] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[5,1] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[0,2] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[1,2] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[2,2] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[3,2] | 1e-05 | 1e-05 | 0.00316228 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | 1 |
| prob_b_comparison[4,2] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[5,2] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[0,3] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[1,3] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[2,3] | 0.99999 | 1e-05 | 0.00316228 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | 1 |
| prob_b_comparison[3,3] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[4,3] | 0.62121 | 0.00153398 | 0.485088 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 100000 | 0.999983 |
| prob_b_comparison[5,3] | 0.00526 | 0.000240106 | 0.0723352 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 90760 | 0.999992 |
| prob_b_comparison[0,4] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[1,4] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[2,4] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[3,4] | 0.37879 | 0.00153398 | 0.485088 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 100000 | 0.999983 |
| prob_b_comparison[4,4] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| prob_b_comparison[5,4] | 0.00256 | 0.000167342 | 0.0505319 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 91184 | 1 |
| prob_b_comparison[0,5] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[1,5] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[2,5] | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 100000 | nan |
| prob_b_comparison[3,5] | 0.99474 | 0.000240106 | 0.0723352 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 90760 | 0.999992 |
| prob_b_comparison[4,5] | 0.99744 | 0.000167342 | 0.0505319 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 91184 | 1 |
| prob_b_comparison[5,5] | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 100000 | nan |
| log_lik[0] | -2.73633 | 0.000935194 | 0.295734 | -3.43849 | -3.28893 | -2.90328 | -2.68784 | -2.5182 | -2.34766 | -2.30554 | 100000 | 1.00002 |
| log_lik[1] | -2.53453 | 0.00070078 | 0.221606 | -3.08005 | -2.95548 | -2.65052 | -2.49078 | -2.37503 | -2.25745 | -2.22527 | 100000 | 0.999975 |
| log_lik[2] | -3.34177 | 0.00146612 | 0.463629 | -4.39499 | -4.1848 | -3.6203 | -3.28603 | -3.00338 | -2.6859 | -2.6025 | 100000 | 0.999994 |
| log_lik[3] | -4.21624 | 0.00207097 | 0.654899 | -5.66336 | -5.38642 | -4.62001 | -4.15503 | -3.74499 | -3.25356 | -3.11148 | 100000 | 0.999974 |
| log_lik[4] | -2.73634 | 0.000936502 | 0.296148 | -3.44194 | -3.28963 | -2.90404 | -2.68719 | -2.51799 | -2.34749 | -2.30663 | 100000 | 0.999975 |
| log_lik[5] | -2.73634 | 0.000936502 | 0.296148 | -3.44194 | -3.28963 | -2.90404 | -2.68719 | -2.51799 | -2.34749 | -2.30663 | 100000 | 0.999975 |
| log_lik[6] | -2.33272 | 0.000426818 | 0.111143 | -2.59392 | -2.52945 | -2.39027 | -2.31979 | -2.25852 | -2.17872 | -2.15482 | 67807 | 1.0002 |
| log_lik[7] | -2.33272 | 0.000426818 | 0.111143 | -2.59392 | -2.52945 | -2.39027 | -2.31979 | -2.25852 | -2.17872 | -2.15482 | 67807 | 1.0002 |
| log_lik[8] | -2.33272 | 0.000426818 | 0.111143 | -2.59392 | -2.52945 | -2.39027 | -2.31979 | -2.25852 | -2.17872 | -2.15482 | 67807 | 1.0002 |
| log_lik[9] | -2.53452 | 0.00069946 | 0.221189 | -3.07629 | -2.95565 | -2.65031 | -2.49165 | -2.37471 | -2.25725 | -2.22585 | 100000 | 1.00006 |
| log_lik[10] | -2.54659 | 0.000785481 | 0.248391 | -3.16542 | -3.02514 | -2.67084 | -2.4917 | -2.36823 | -2.24892 | -2.2172 | 100000 | 1.00005 |
| log_lik[11] | -2.34498 | 0.000496465 | 0.125425 | -2.64993 | -2.57005 | -2.40342 | -2.3269 | -2.26265 | -2.18144 | -2.15687 | 63825 | 1.00009 |
| log_lik[12] | -2.34498 | 0.000496465 | 0.125425 | -2.64993 | -2.57005 | -2.40342 | -2.3269 | -2.26265 | -2.18144 | -2.15687 | 63825 | 1.00009 |
| log_lik[13] | -2.74833 | 0.00104876 | 0.331647 | -3.546 | -3.37344 | -2.93064 | -2.68727 | -2.50315 | -2.32712 | -2.28515 | 100000 | 1.00003 |
| log_lik[14] | -2.41232 | 0.000613607 | 0.176642 | -2.86322 | -2.75229 | -2.4868 | -2.3749 | -2.29388 | -2.20328 | -2.1765 | 82872 | 1.00003 |
| log_lik[15] | -3.01794 | 0.00133705 | 0.422813 | -4.00776 | -3.80561 | -3.25894 | -2.95459 | -2.70599 | -2.4528 | -2.39164 | 100000 | 1.00002 |
| log_lik[16] | -2.74833 | 0.00104876 | 0.331647 | -3.546 | -3.37344 | -2.93064 | -2.68727 | -2.50315 | -2.32712 | -2.28515 | 100000 | 1.00003 |
| log_lik[17] | -2.54692 | 0.000791085 | 0.250163 | -3.16951 | -3.02844 | -2.67035 | -2.49157 | -2.36771 | -2.24974 | -2.21829 | 100000 | 1.00002 |
| log_lik[18] | -3.73765 | 0.00205657 | 0.650345 | -5.21599 | -4.92322 | -4.1275 | -3.66109 | -3.2632 | -2.81517 | -2.69676 | 100000 | 0.999991 |
| log_lik[19] | -2.5447 | 0.000828546 | 0.262009 | -3.21331 | -3.05791 | -2.67094 | -2.48239 | -2.35768 | -2.24115 | -2.21039 | 100000 | 1.00001 |
| log_lik[20] | -2.42805 | 0.000693641 | 0.195391 | -2.93941 | -2.809 | -2.50779 | -2.38289 | -2.29826 | -2.2048 | -2.17837 | 79349 | 1.00005 |
| log_lik[21] | -2.3563 | 0.000560908 | 0.138402 | -2.70641 | -2.61165 | -2.4159 | -2.33316 | -2.26661 | -2.1839 | -2.15899 | 60884 | 1.00014 |
| log_lik[22] | -4.81377 | 0.00283885 | 0.897723 | -6.79217 | -6.42019 | -5.36376 | -4.72578 | -4.16988 | -3.49737 | -3.30988 | 100000 | 0.999977 |
| log_lik[23] | -2.41463 | 0.000656152 | 0.184738 | -2.90055 | -2.776 | -2.48901 | -2.37315 | -2.29235 | -2.20083 | -2.17451 | 79269 | 1.00006 |
| log_lik[24] | -2.35183 | 0.000548798 | 0.133508 | -2.68773 | -2.59816 | -2.41028 | -2.33023 | -2.26506 | -2.18237 | -2.15774 | 59182 | 1.00015 |
| log_lik[25] | -3.68089 | 0.00179969 | 0.569113 | -4.95951 | -4.71565 | -4.02176 | -3.61904 | -3.26868 | -2.86285 | -2.7564 | 100000 | 0.99997 |
| log_lik[26] | -2.39015 | 0.000531975 | 0.156079 | -2.78711 | -2.69127 | -2.45852 | -2.36012 | -2.28619 | -2.19753 | -2.17168 | 86081 | 1.00001 |
| log_lik[27] | -2.70464 | 0.000949753 | 0.300338 | -3.42914 | -3.27577 | -2.86761 | -2.65075 | -2.48359 | -2.32096 | -2.28113 | 100000 | 0.999965 |
| log_lik[28] | -2.70464 | 0.000949753 | 0.300338 | -3.42914 | -3.27577 | -2.86761 | -2.65075 | -2.48359 | -2.32096 | -2.28113 | 100000 | 0.999965 |
| log_lik[29] | -2.3338 | 0.000448643 | 0.114007 | -2.60405 | -2.53915 | -2.39156 | -2.31964 | -2.25791 | -2.17838 | -2.15378 | 64574 | 1.00008 |
| log_lik[30] | -2.56839 | 0.000779171 | 0.246395 | -3.17826 | -3.04225 | -2.69622 | -2.51769 | -2.39041 | -2.2649 | -2.23265 | 100000 | 0.999982 |
| log_lik[31] | -4.30475 | 0.00222789 | 0.70452 | -5.86668 | -5.56526 | -4.73925 | -4.23866 | -3.80034 | -3.26551 | -3.12075 | 100000 | 0.999971 |
| log_lik[32] | -2.51376 | 0.000705027 | 0.222949 | -3.07066 | -2.94619 | -2.62376 | -2.46574 | -2.35573 | -2.24338 | -2.21269 | 100000 | 0.999976 |
| log_lik[33] | -3.82294 | 0.00190039 | 0.600955 | -5.17464 | -4.90752 | -4.18988 | -3.75953 | -3.39001 | -2.94919 | -2.83484 | 100000 | 0.999968 |
| log_lik[34] | -2.72106 | 0.00102137 | 0.322986 | -3.50706 | -3.33946 | -2.89458 | -2.66028 | -2.48187 | -2.31426 | -2.27516 | 100000 | 0.99998 |
| log_lik[35] | -2.34996 | 0.000499418 | 0.129235 | -2.67219 | -2.58849 | -2.40913 | -2.32994 | -2.2649 | -2.18353 | -2.15836 | 66963 | 1.00008 |
| log_lik[36] | -2.42536 | 0.000583436 | 0.184499 | -2.9031 | -2.7866 | -2.50546 | -2.38443 | -2.3008 | -2.20712 | -2.18051 | 100000 | 1.00001 |
| log_lik[37] | -2.77796 | 0.00108888 | 0.344334 | -3.61273 | -3.42961 | -2.96609 | -2.71626 | -2.52376 | -2.33709 | -2.29548 | 100000 | 0.99997 |
| log_lik[38] | -2.56802 | 0.000821847 | 0.259891 | -3.21964 | -3.07226 | -2.69821 | -2.51079 | -2.37974 | -2.25574 | -2.22447 | 100000 | 0.999978 |
| log_lik[39] | -2.40097 | 0.000587076 | 0.168242 | -2.83704 | -2.72648 | -2.47175 | -2.36656 | -2.28945 | -2.19963 | -2.17254 | 82126 | 1.00004 |
| log_lik[40] | -3.7057 | 0.00191992 | 0.607131 | -5.07782 | -4.81287 | -4.07006 | -3.63623 | -3.26529 | -2.83716 | -2.72761 | 100000 | 0.999971 |
| log_lik[41] | -2.56802 | 0.000821847 | 0.259891 | -3.21964 | -3.07226 | -2.69821 | -2.51079 | -2.37974 | -2.25574 | -2.22447 | 100000 | 0.999978 |
| log_lik[42] | -2.64915 | 0.000989698 | 0.31297 | -3.42632 | -3.25123 | -2.80993 | -2.57989 | -2.41906 | -2.27508 | -2.24037 | 100000 | 0.999983 |
| log_lik[43] | -2.64915 | 0.000989698 | 0.31297 | -3.42632 | -3.25123 | -2.80993 | -2.57989 | -2.41906 | -2.27508 | -2.24037 | 100000 | 0.999983 |
| log_lik[44] | -2.48078 | 0.000720873 | 0.22796 | -3.07134 | -2.92787 | -2.58129 | -2.42439 | -2.32413 | -2.22039 | -2.19265 | 100000 | 0.999998 |
| log_lik[45] | -2.47998 | 0.0007191 | 0.227399 | -3.06829 | -2.92811 | -2.58068 | -2.42439 | -2.32295 | -2.2199 | -2.19207 | 100000 | 0.999991 |
| log_lik[46] | -2.37927 | 0.000572994 | 0.1578 | -2.78904 | -2.68228 | -2.44339 | -2.34819 | -2.27626 | -2.19009 | -2.16486 | 75843 | 1.00002 |
| log_lik[47] | -3.1857 | 0.00158996 | 0.502788 | -4.36319 | -4.12359 | -3.47549 | -3.11292 | -2.81382 | -2.50661 | -2.43336 | 100000 | 0.999983 |
| log_lik[48] | -2.34584 | 0.00051513 | 0.126888 | -2.65972 | -2.57614 | -2.40448 | -2.32682 | -2.26275 | -2.18111 | -2.15621 | 60675 | 1.00006 |
| log_lik[49] | -2.41088 | 0.000514388 | 0.162664 | -2.82099 | -2.72195 | -2.48676 | -2.37974 | -2.29996 | -2.20902 | -2.18182 | 100000 | 0.999999 |
| log_lik[50] | -2.76093 | 0.000962221 | 0.304281 | -3.48081 | -3.32983 | -2.932 | -2.71175 | -2.53689 | -2.35856 | -2.31605 | 100000 | 0.999983 |
| log_lik[51] | -2.55227 | 0.000724273 | 0.229035 | -3.11222 | -2.98866 | -2.67227 | -2.5078 | -2.38647 | -2.26473 | -2.23413 | 100000 | 0.999982 |
| log_lik[52] | -2.32991 | 0.000421662 | 0.109997 | -2.58737 | -2.52469 | -2.38765 | -2.31737 | -2.25642 | -2.17761 | -2.15289 | 68051 | 1.00014 |
| log_lik[53] | -2.97519 | 0.001168 | 0.369353 | -3.83217 | -3.65693 | -3.18955 | -2.92343 | -2.70504 | -2.47099 | -2.41237 | 100000 | 1.00001 |
| log_lik[54] | -2.32991 | 0.000421662 | 0.109997 | -2.58737 | -2.52469 | -2.38765 | -2.31737 | -2.25642 | -2.17761 | -2.15289 | 68051 | 1.00014 |
| log_lik[55] | -2.41088 | 0.000514388 | 0.162664 | -2.82099 | -2.72195 | -2.48676 | -2.37974 | -2.29996 | -2.20902 | -2.18182 | 100000 | 0.999999 |
| log_lik[56] | -2.76093 | 0.000962221 | 0.304281 | -3.48081 | -3.32983 | -2.932 | -2.71175 | -2.53689 | -2.35856 | -2.31605 | 100000 | 0.999983 |
| log_lik[57] | -2.55227 | 0.000724273 | 0.229035 | -3.11222 | -2.98866 | -2.67227 | -2.5078 | -2.38647 | -2.26473 | -2.23413 | 100000 | 0.999982 |
| log_lik[58] | -5.96118 | 0.00310773 | 0.982751 | -8.08284 | -7.69382 | -6.58038 | -5.88692 | -5.26594 | -4.47592 | -4.2481 | 100000 | 1 |
| log_lik[59] | -2.52657 | 0.000757293 | 0.239477 | -3.12965 | -2.99042 | -2.64358 | -2.47232 | -2.35648 | -2.24149 | -2.21098 | 100000 | 0.999994 |
| log_lik[60] | -2.42542 | 0.000581973 | 0.184036 | -2.89803 | -2.78347 | -2.50676 | -2.38427 | -2.30052 | -2.2072 | -2.17959 | 100000 | 1.00003 |
| log_lik[61] | -3.70403 | 0.00191809 | 0.606552 | -5.07289 | -4.8054 | -4.06898 | -3.63492 | -3.26146 | -2.84054 | -2.73181 | 100000 | 1 |
| log_lik[62] | -2.34145 | 0.000491539 | 0.121126 | -2.63625 | -2.55886 | -2.40019 | -2.32462 | -2.26172 | -2.18046 | -2.15654 | 60724 | 1.00006 |
| log_lik[63] | -2.3498 | 0.000495617 | 0.128819 | -2.66905 | -2.58578 | -2.40933 | -2.33002 | -2.26458 | -2.18315 | -2.158 | 67557 | 1.00007 |
| log_lik[64] | -2.34145 | 0.000491539 | 0.121126 | -2.63625 | -2.55886 | -2.40019 | -2.32462 | -2.26172 | -2.18046 | -2.15654 | 60724 | 1.00006 |
| log_lik[65] | -3.05587 | 0.00137053 | 0.4334 | -4.06844 | -3.86188 | -3.3063 | -2.9924 | -2.73696 | -2.46901 | -2.40783 | 100000 | 0.999993 |
| log_lik[66] | -2.5683 | 0.000819629 | 0.259189 | -3.21226 | -3.06855 | -2.70014 | -2.51147 | -2.38054 | -2.25607 | -2.22414 | 100000 | 1.00001 |
| log_lik[67] | -2.49983 | 0.00075334 | 0.238227 | -3.10952 | -2.96599 | -2.60912 | -2.4412 | -2.33334 | -2.22697 | -2.19767 | 100000 | 1 |
| log_lik[68] | -2.36999 | 0.00057377 | 0.149574 | -2.75292 | -2.65382 | -2.43233 | -2.34244 | -2.27251 | -2.18818 | -2.16267 | 67957 | 1.00001 |
| log_lik[69] | -2.36999 | 0.00057377 | 0.149574 | -2.75292 | -2.65382 | -2.43233 | -2.34244 | -2.27251 | -2.18818 | -2.16267 | 67957 | 1.00001 |
| log_lik[70] | -2.36999 | 0.00057377 | 0.149574 | -2.75292 | -2.65382 | -2.43233 | -2.34244 | -2.27251 | -2.18818 | -2.16267 | 67957 | 1.00001 |
| log_lik[71] | -2.46125 | 0.000725389 | 0.215494 | -3.01841 | -2.88701 | -2.5549 | -2.4086 | -2.31443 | -2.21489 | -2.1868 | 88253 | 0.999968 |
| log_lik[72] | -2.38928 | 0.000607463 | 0.165664 | -2.81825 | -2.70878 | -2.45625 | -2.35539 | -2.28072 | -2.19397 | -2.16786 | 74373 | 1.00004 |
| log_lik[73] | -2.38928 | 0.000607463 | 0.165664 | -2.81825 | -2.70878 | -2.45625 | -2.35539 | -2.28072 | -2.19397 | -2.16786 | 74373 | 1.00004 |
| log_lik[74] | -3.67851 | 0.0018051 | 0.570824 | -4.96139 | -4.70864 | -4.02303 | -3.61492 | -3.26414 | -2.86209 | -2.75324 | 100000 | 0.999999 |
| log_lik[75] | -2.42412 | 0.000559944 | 0.17707 | -2.87225 | -2.76381 | -2.50505 | -2.38741 | -2.30404 | -2.20929 | -2.18255 | 100000 | 1.00006 |
| log_lik[76] | -4.85816 | 0.00258955 | 0.818888 | -6.64133 | -6.31093 | -5.36921 | -4.78662 | -4.26925 | -3.64582 | -3.46943 | 100000 | 0.999995 |
| log_lik[77] | -2.39 | 0.000493201 | 0.155964 | -2.78444 | -2.6866 | -2.4595 | -2.36016 | -2.28576 | -2.1976 | -2.17153 | 100000 | 1 |
| log_lik[78] | -2.9613 | 0.00121791 | 0.385138 | -3.86024 | -3.67563 | -3.1825 | -2.90355 | -2.6775 | -2.44311 | -2.38634 | 100000 | 0.999986 |
| log_lik[79] | -2.39 | 0.000493201 | 0.155964 | -2.78444 | -2.6866 | -2.4595 | -2.36016 | -2.28576 | -2.1976 | -2.17153 | 100000 | 1 |
| log_lik[80] | -3.41139 | 0.00160314 | 0.506957 | -4.55559 | -4.33322 | -3.71439 | -3.35026 | -3.03988 | -2.69708 | -2.60639 | 100000 | 0.999997 |
| log_lik[81] | -3.67851 | 0.0018051 | 0.570824 | -4.96139 | -4.70864 | -4.02303 | -3.61492 | -3.26414 | -2.86209 | -2.75324 | 100000 | 0.999999 |
| log_lik[82] | -2.78322 | 0.00103984 | 0.328827 | -3.56402 | -3.39689 | -2.96697 | -2.72775 | -2.54094 | -2.35577 | -2.31086 | 100000 | 1.00001 |
| log_lik[83] | -2.84325 | 0.0011636 | 0.367961 | -3.72012 | -3.53811 | -3.04991 | -2.78014 | -2.57055 | -2.36481 | -2.31905 | 100000 | 1 |
| log_lik[84] | -2.36323 | 0.000525796 | 0.140284 | -2.71569 | -2.62599 | -2.42459 | -2.33932 | -2.27104 | -2.18727 | -2.16231 | 71184 | 0.999996 |
| log_lik[85] | -2.66497 | 0.000953673 | 0.301578 | -3.40396 | -3.24526 | -2.82653 | -2.60503 | -2.44195 | -2.29206 | -2.25465 | 100000 | 1.00001 |
| log_lik[86] | -2.37956 | 0.000542134 | 0.152967 | -2.77117 | -2.67185 | -2.44493 | -2.35106 | -2.27869 | -2.19225 | -2.16674 | 79613 | 1 |
| log_lik[87] | -2.37956 | 0.000542134 | 0.152967 | -2.77117 | -2.67185 | -2.44493 | -2.35106 | -2.27869 | -2.19225 | -2.16674 | 79613 | 1 |
| log_lik[88] | -2.37956 | 0.000542134 | 0.152967 | -2.77117 | -2.67185 | -2.44493 | -2.35106 | -2.27869 | -2.19225 | -2.16674 | 79613 | 1 |
| log_lik[89] | -2.61597 | 0.000890215 | 0.281511 | -3.30934 | -3.15762 | -2.7638 | -2.55755 | -2.40974 | -2.27316 | -2.23913 | 100000 | 1 |
| log_lik[90] | -2.37956 | 0.000542134 | 0.152967 | -2.77117 | -2.67185 | -2.44493 | -2.35106 | -2.27869 | -2.19225 | -2.16674 | 79613 | 1 |
| log_lik[91] | -2.35169 | 0.000524891 | 0.13297 | -2.6827 | -2.595 | -2.41106 | -2.33035 | -2.26492 | -2.18286 | -2.1578 | 64175 | 0.999993 |
| log_lik[92] | -3.39297 | 0.00177845 | 0.562394 | -4.69445 | -4.4338 | -3.71977 | -3.31813 | -2.98064 | -2.61791 | -2.52666 | 100000 | 0.999975 |
| log_lik[93] | -2.56962 | 0.000873156 | 0.276116 | -3.26589 | -3.1083 | -2.7045 | -2.50481 | -2.37047 | -2.24856 | -2.21752 | 100000 | 0.999975 |
| log_lik[94] | -2.73918 | 0.00110666 | 0.349957 | -3.59245 | -3.40636 | -2.92461 | -2.66867 | -2.48011 | -2.3068 | -2.26847 | 100000 | 0.999968 |
| log_lik[95] | -2.35169 | 0.000524891 | 0.13297 | -2.6827 | -2.595 | -2.41106 | -2.33035 | -2.26492 | -2.18286 | -2.1578 | 64175 | 0.999993 |
| log_lik[96] | -3.73209 | 0.00205986 | 0.651384 | -5.20444 | -4.91743 | -4.1244 | -3.65239 | -3.25571 | -2.80926 | -2.69637 | 100000 | 0.999975 |
| log_lik[97] | -2.56962 | 0.000873156 | 0.276116 | -3.26589 | -3.1083 | -2.7045 | -2.50481 | -2.37047 | -2.24856 | -2.21752 | 100000 | 0.999975 |
水準の差
| changeup | curve | cutball | fork | slider | straight | |
|---|---|---|---|---|---|---|
| changeup | 0 | 0.99474 | 0 | 1e-05 | 0.37879 | 0 |
| curve | 0.00526 | 0 | 0 | 0 | 0.00256 | 0 |
| cutball | 1 | 1 | 0 | 1 | 1 | 0 |
| fork | 0.99999 | 1 | 0 | 0 | 1 | 0 |
| slider | 0.62121 | 0.99744 | 0 | 0 | 0 | 0 |
| straight | 1 | 1 | 1 | 1 | 1 | 0 |

考察
- 水準の効果の分布より、走者の状況も交絡因子も殆ど影響力が無いことが推し量れる
- 説明量や効果量についても、圧倒的に走者(b)についての値が大きく、モデルの殆どがこれで説明される事がわかる
- 交絡因子の大きさについても図れるのはメリットぽい。